7 KARAKTER MOLEKULER Chili veinal mottle virus ISOLAT LEMAH (Molecular Characterization of Weak Isolates of Chili veinal mottle virus)
|
|
|
- Lanny Tan
- 6 tahun lalu
- Tontonan:
Transkripsi
1 55 7 AKTER MOLEKULER Chili veinal mottle virus ISOLAT LEMAH (Molecular Characterization of Weak Isolates of Chili veinal mottle virus) Abstrak Pre-imunisasi dengan isolat-isolat lemah Chili veinal mottle virus (,,,, dan ) efektif melindungi tanaman cabai dari infeksi isolat kuat ChiVMV-CKB. Analisis gen coat protein (CP) dari 5 ChiVMV isolat lemah tersebut dilakukan melalui perunutan sikuen nukleotida dan asam amino, dilanjutkan dengan penyejajaran (alligment) sikuen dan analisis filogeni. Di antara kelima ChiVMV isolat lemah ditemukan keragaman sikuen nukleotida dan asam amino, tetapi analisis filogenetik menunjukkan bahwa kelima ChiVMV isolat lemah memiliki tingkat homologi yang tinggi dan berada dalam satu kelompok dengan isolat kuat ChiVMV-CKB. Hasil analisis karakter molekuler ChiVMV isolat lemah tersebut menjelaskan keefektivan kelima isolat lemah sebagai agens proteksi silang. Kata kunci : Chili veinal mottle virus, kesamaan, nukleotida, sikuen Abstract Pre-immunization with weak isolates of Chili veinal mottle virus (,,,, and ) was able to protect chili pepper from infection of severe isolate ChiVMV-CKB. Analysis of coat protein (CP) gene was done by sequencing, followed by allignment and phyllogenetic analysis. Sequence variability was found among 5 weak isolates, however phyllogenetic analysis showed that all weak isolates has high sequence homology and belong to the same group with severe isolate ChiVMV-CKB. This molecular characterization of weak isolates of ChiVMV explained their effectiveness as agents for cross protection against infection of severe isolates of ChiVMV. Keywords : Chili veinal mottle virus, homolog, nucleotides, sequences
2 56 Pendahuluan Chili veinal mottle virus (ChiVMV) merupakan salah satu anggota Potyvirus yang menginfeksi cabai, menimbulkan gejala belang hijau tua, malformasi daun, dan pengurangan ukuran daun (Lee et al. 2005), buah lebih sedikit dan lebih kecil (Ong et al 1979). Virus tersebut tersebar luas di Asia (Green dan Kim 1991; Tsai et al. 2008; Wang et al. 2006). Karakterisasi molekuler ChiVMV yang berasal dari daerah dan negara yang berbeda telah dilakukan, umumnya melalui analisis susunan sikuen nukleotida serta asam amino dari gen coat protein (CP). Chiemsombat et al. (1998) berhasil mengamplifkasi gen CP ChiVMV isolat Thailand dengan teknik reverse transcription polymeration chain reaction (RT-PCR). Moury et al. (2005) mendeteksi 2 spesies Potyvirus yaitu ChiVMV dan Pepper veinal mottle virus (PVMV) berdasarkan analisis sikuen gen CP yang diperoleh dengan teknik RT- PCR. Selanjutnya Tsai et al. (2008) melaporkan hasil amplifikasi gen CP 24 isolat ChiVMV yang berasal dari Taiwan, Thailand, India, China, dan Indonesia. Pasangan primer yang digunakan untuk mengamplifikasi gen CP isolat ChiVMV dari Indonesia (Pata-F1/R1) berbeda dengan pasangan primer yang digunakan untuk mengamplifikasi gen CP isolat ChiVMV dari negara lainnya (Poty 3 dan Oligo [dt]). Hal tersebut menjadi indikasi adanya perbedaan gen CP isolat ChiVMV dari Indonesia. Analisis sikuen gen CP ChiVMV menunjukkan tingkat homologi yang beragam. Lee et al. (2005) melaporkan gen CP ChiVMV memiliki kesamaan dengan ChiVMV isolat India (kode GeneBank Nc ) sebesar 87.1% dan 91.7% berturut-turut berdasarkan sikuen nukleotida dan asam amino. Moury et al. (2005) mempelajari hubungan kekerabatan 3 isolat ChiVMV dari Taiwan, Beijing, Thailand dan melaporkan bahwa ketiga isolat tersebut memiliki tingkat kesamaan sikuen nukleotida 90.3% sampai 92.8%. Analisis sikuen gen CP ChiVMV yang dilakukan oleh Tsai et al. (2008) menunjukkan bahwa 24 isolat ChiVMV dari Taiwan, Thailand, India, China, dan Indonesia memiliki tingkat kesamaan nukleotida dan asam amino berturut-turut 88.6% sampai 99.9% dan 95.1% sampai 100%. Berdasarkan analisis filogenetik sikuen nukleotida gen CP maka 24 isolat ChiVMV dikelompokkan menjadi 3 kelompok, kelompok pertama adalah semua isolat dari China dan Taiwan, 3 isolat dari Thailand, kelompok kedua adalah semua isolat dari Indonesia, 5 isolat dari Thailand, dan Chili vein-banding mottle virus (CVbMV), dan kelompok ketiga adalah semua isolat dari India dan Pepper vein-banding virus (PVBV) (Tsai et al. 2008). Karakterisasi molekuler ChiVMV yang telah dilaporkan umumnya berkaitan dengan ChiVMV isolat kuat yang menyebabkan penyakit pada tanaman cabai. Karakterisasi ChiVMV isolat lemah perlu dilakukan berkaitan dengan pemanfaatannya sebagai agens proteksi silang. Salah satu mekanisme proteksi silang adalah post transcriptional gene silencing (PTGS). Mekanisme ini sangat berkaitan dengan tingginya tingkat kesamaan sikuen antara isolat lemah dengan isolat kuat. Pada penelitian ini dilaporkan analisis sikuen gen CP dari 5 ChiVMV isolat lemah (,,,, dan ) yang potensial sebagai agens proteksi silang. Karakteristik molekuler ChiVMV isolat lemah didasarkan pada sikuen gen CP yang akan
3 57 menjadi informasi dasar untuk pengembangan strategi pengendalian penyakit melalui teknik proteksi silang. Bahan dan Metode Tempat dan Waktu Penelitian Penelitian dilakukan di Rumah Kaca Cikabayan dan Laboratorium Virologi Tumbuhan Departemen Proteksi Tanaman, Fakultas Pertanian, Institut Pertanian Bogor, dari bulan Desember 2011 sampai bulan April Isolat ChiVMV Lima isolat-isolat lemah ChiVMV yang digunakan pada penelitian ini adalah isolat,,,, dan yang merupakan hasil penelitian sebelumnya ( Interaksi antara Chili veinal mottle virus isolat lemah dengan Isolat Kuat [BAB 5]). ChiVMV isolat lemah masing-masing diinokulasi secara mekanis ke tanaman cabai C.annuum IPB C13. Tanaman yang telah diinokulasi dipelihara dalam rumah kaca kedap serangga, selanjutnya digunakan untuk ekstraksi RNA. Deteksi ChiVMV Menggunakan Metode RT-PCR Metode RT-PCR terdiri atas 3 tahapan yaitu ekstraksi RNA total, reaksi transkripsi balik (reverse transcription), dan amplifikasi cdna. Ekstraksi RNA total Ekstraksi RNA total dilakukan menurut metode Xprep dengan menggunakan Xprep Plant RNA Mini Kit (Phile, Korea). Sebanyak 0.1 g daun tanaman cabai yang terinfeksi digerus menggunakan pistil dan mortar yang sebelumnya diberi nitrogen cair hingga menjadi bubuk. Sebanyak 450 μl bufer XPRB yang telah ditambah 4.5 μl merkaptoetanol dimasukkan kedalam hasil gerusan. Sap yang dihasilkan dimasukkan kedalam tabung mikro 1.5 ml. Sap dipindahkan kedalam filter kolom putih, disentrifugasi dengan kecepatan rpm selama 2 menit pada suhu kamar. Supernatan diambil dan dipindahkan ke tabung berukuran 2 ml, selanjutnya diberi 0.5 volume etanol 96% dan dicampurkan hingga homogen, kemudian dipindahkan ke XPPLR mini kolom merah, disentrifugasi dengan kecepatan rpm selama 1 menit. XPPLR mini kolom merah ditempatkan diatas tabung lalu ditambahkan 500 μl wash buffer 1, selanjutnya di sentrifugasi pada kecepatan rpm selama 1 menit. Selanjutnya XPPLR mini kolom merah ditempatkan diatas tabung dan ditambahkan 750 μl wash buffer 2, disentrifugasi pada kecepatan rpm selama 1 menit dan diulangi lagi 3 menit untuk mengeringkan kolom. XPPLR mini kolom merah dimasukkan lagi ke tabung ditambahkan 50 μl water free nuclease, didiamkan 1 menit, di sentrifugasi pada kecepatan rpm selama 2 menit. Hasil ekstraksi RNA total disimpan pada suhu -80 o C sampai akan digunakan.
4 58 RT-PCR Tabung mikro diisi dengan 25 μl reaksi transkripsi balik terdiri atas 7.5 μl water free nuclease, 5 μl bufer RT 5x, 5 μl dntp 10 mm, oligo d (T) 10 μm, μl Rnase inhibitor, μl MmLV Rpv, μl dtt, 3 μl RNA sampel. Tabung mikro dimasukkan ke dalam thermal cycler ( Gene Amp PCR System 9700) dengan kondisi RT suhu 25 o C selama 5 menit, 37 o C selama 90 menit dan 70 o C selama 15 menit. Hasil akhir adalah produk cdna. Amplifikasi cdna Amplifikasi cdna ChiVMV pada daerah coat protein (CP) menggunakan pasangan primer ChiVMV F Ind (1 st BASE) (5 AACCTGAGCGTATAGTTTCA 3 ) dan ChiVMV R Ind (1 st BASE) (5 TACGCTTCAGCAAGATTGCT 3 ) (Tsai et al. 2008). Tabung mikro diisi dengan reaksi amplifikasi 25 μl terdiri atas 8.5 μl water free nuclease, 12.5 μl 2x kappa master mix, 1 μl primer ChiVMV F Ind, 1 μl primer ChiVMV R Ind, 2 μl cdna dari RT. Tabung mikro dimasukkan ke dalam thermal cycler ( Gene Amp PCR System 9700). Amplifikasi ChiVMV sebanyak 35 siklus yaitu pemisahan utas DNA pada suhu 94 o C selama 1 menit, penempelan primer pada DNA pada suhu 47 o C selama 1 menit dan sintesis DNA pada suhu 72 o C selama 2 menit, untuk siklus terakhir ditambah tahapan sintesis selama 15 menit, siklus berakhir pada suhu 4 o C. Fragmen DNA hasil amplifikasi diamati secara elektroforesis pada agarosa 1% dalam larutan penyangga Tris-buffer EDTA (TBE 0.5x), dengan kekuatan 100 volt selama 20 menit. Ukuran produk amplifikasi PCR diperkirakan dengan pembanding menggunakan DNA standar (1 kb ladder), selanjutnya diamati menggunakan sinar UV setelah diberi etidium bromida (10 mg/l). Jika amplifikasi PCR berhasil akan diperoleh fragmen DNA dengan ukuran 900 pasang basa (Tsai et al. 2005). Perunutan dan Analisis Sikuen Gen CP ChiVMV Hasil amplifikasi sampel DNA digunakan untuk mengetahui sikuen nukleotida. Perunutan DNA semua sampel ChiVMV isolat lemah dilakukan di Laboratorium Genetica Science di Singapore. Hasil perunutan selanjutnya digunakan untuk analisis keragaman berdasarkan tingkat kesamaan sikuen nukleotida. Sikuen nukleotida ChiVMV dibandingkan dengan sikuen nukleotida ChiVMV yang tersimpan di GeneBank menggunakan software Blast2.Wu ( Analisis similaritas dilakukan dengan membandingkan homologi gen CP ChiVMV isolat lemah dengan 9 isolat ChiVMV dari beberapa negara dan 1 isolat CTV (sebagai out group) (Tabel 7.1) Matriks identitas nukleotida dan asam amino diperoleh dengan mengunakan software Bioedit. Analisis filogenetika menggunakan fasilitas yang tersedia pada
5 59 Tabel 7.1 Sikuen nukleotida Chili veinal mottle virus yang berasal dari GeneBank yang digunakan sebagai pembanding pada analisis keragaman isolatisolat lemah ChiVMV No Isolat Daerah asal Kode aksesi Aksesor* CKB Pataruman Wenchang S7 HCH9 D10 Ca CM1 VN/C2 CTV-8 Indonesia Indonesia China India India India Korea Selatan Thailand Vietnam Portugal DQ DQ GQ EF EF EF AJ AB DQ JQ Tsai et al. 2008*** Tsai et al. 2008*** Wu et al. 2009** Reddy et al. 2007** Reddy et al. 2007** Reddy et al. 2007** Yoon et al. 2008** Ikegami et al. 1999*** Ha et al. 2008*** Cerni dan Nelasco 2012** *Aksesor yang mendaftarkan sikuen isolat ChiVMV ke GeneBank; - tidak terdaftar di GeneBank, ** tidak dipublikasikan pada jurnal; *** dipublikasikan pada jurnal. Hasil dan Pembahasan Amplifikasi DNA dan Perunutan Nukleotida Gen CP Pita DNA berukuran 900 bp berhasil teramplifikasi dari 5 ChiVMV isolat lemah dan 1 isolat kuat ChiVMV-CKB menggunakan pasangan primer ChiVMV F Ind dan ChiVMV R Ind (Gambar 7.1). Setelah dilakukan perunutan nukleotida diperoleh urutan nukleotida sepanjang 913, 907, 907, 905, dan 906 berturut-turut untuk isolat (, SPT,,, dan ) (Gambar 7.2) M bp Gambar 7.1 Visualisasi pita DNA hasil amplifikasi dengan primer ChiVMV F Ind dan ChiVMV R Ind pada gel agarosa TBE; M) marker (1 kb ladder), 1) tanaman sehat, 2), 3), 4), 5) CKB, 6), dan 7).
6 AACCTGAGGCGTATAGTTTCAATTCTAGACTGAGGGCAAGACTGTACAACCAGAACACCGACTTGAAGCC AACCTGAAGCGTATAGTTTCAATTCTTGAATG-GGATAGAGCTGAACAGCCAGAACATCGACTTGAAGCT AACCTGAGGCGTATAGTTTCAATTCTTGAGTG-GGATAGAGCTGAACAGCCAGAACATCGACTTGAAGCT AACCTGAGGCGTATAGTTTCAATTCTAGAATG-GGATAGGGCTGAACAACCAGAACACCGACTTGAAGCC AACCTGAAGCGTATAGTTTCAATTCTAGAATG-GGATAGGGCTGAAAAACCAGAACACCGACTTGAAGCC AACCTGA-GCGTATAGTTTCAATTCTAGAATG-GGATAGGGCTGAACAACCAGAACACCGACTTGAAGCC ATTTGTGCATCTATGATTGAAGCGTGGGGTTACAAGGAACTTTTACATGAGATAAGGCTTTTCTATAAGT ATTTGTGCATCTATGATTGAAGCATGGGGATACAAAGAACTACTACATGAGATAAGGCTTTTCTATAAAT ATTTGTGCATCTATGATTGAAGCATGGGGTTACAAAGAACTATTACATGAGATAAGGCTTTTCTATAAAT ATTTGTGCATCTATGATTGAAGCGTGGGGTTACAGGGAACTTTTACATGAGATAAGGCTTTTCTATAAGT ATTTGTGCATCTATGATTGAAGCGTGGGGTTACAGGGAACTTTTACATGAGATAAGGCTTTTCTATAAGT ATTTGTGCATCTATGATTGAAGCGTGGGGTTACAAGGAACTTTTACATGAGATAAGGCTTTTCTATAAGT GGGTGATTGAACAAGCACCATACTCACAACTTGTTTCTGAGGGAAAAGCACCATACATTTCAGAAACAGC GGGTGATCGAGCAAGCACCATATTCACAACTTGTCTCTGAGGGAAAGGCACCATACATCTCAGAAACAGC GGGTGATCGAGCAAGCACCATATTCACAACTTGTCTCCGAGGGAAAGGCACCATACATCTCAGAAACAGC GGGTGATTGAACAAGCACCATACTCACAACTTGTTTCTGAGGGAAAAGCACCATACATTTCAGAAACAGC GGGTGATTGAACAAGCACCATACTCACAACTTGTTTCTGAGGGAAAAGCACCATACATTTCAGAAACAGC GGGTGATTGAACAAGCACCATACTCACAACTTGTTTCTGAGGGAAAAGCACCATACATTTCAGAAACAGC ATTGAGATGCCTCTACATGAGTGGACATGGAGAAAGTGATATCAGCCCATATTTGAGAGCACTCATTGAA ACTGAGGTGCCTCTACATGAGTGAGCATGGAGAGAGTGACATTAGTCCATATTTGAGAGCACTCATTGAA ACTGAGGTGCCTCTACATGAGTGAGCATGGAGAGAGTGACATTAGTCCATATTTGAGAGCACTCATTGAA ATTGAGATGTCTCTACATGAGTGAACATGGAGAAAGTGATATCAGCCCATATCTGAGAGCACTTATTGAA ATTGAGATGTCTCTACATGAGTGAACATGGAGAAAGTGATATCAGCCCATATCTGAGAGCACTTATTGAA ATTGAGATGTCTCTACGTGAGTGAACATGGAGAAAGTGATATCAGCCCATATCTGAGAGCACTTATTGAA GGAGCTAGGAGGGAAGAATTAGATGATGAGAGTGGAGAAGTTACCCATCAGTCGGGAGAGAGTGTTGATG GGGGCCAAGGAGGAAGGATTAGATGACGAGGGTGGGGAAGTTGCCCATCAGTCGGGAGAGAGCGTTGATG GGGGCCAAGGAGGAAGGATTAGATGACGAAGGTGGGGAAGTTGCCCATCAGTCAGGAGAGAGCATTGATG GGAGCTAGGAGGGAAGAATTAAATGATGAGGGTGGAGAAGTTACCCATCAGTCGGGAGAAAGTGTTGATG GGAGCTAGGAGGGAAGAATTAAATGATGAGGGTGGAGAAGTTACCCATCAGTCGGGAGAAAGTGTTGATG GGAGCTAGGAGGGAAGAATTAAATGATGAGGGTGGAGAAGTTACCCATCAGTCGGGAGAGAGTGTTGATG CTGGAAGAATAAAAGGTGAAGATTCTTCAAACAAGTCAGCTGATAAACAAGCCACAGATAAGAAGAGCAA CTGGAAGAGTTAAGGGCGAAGACTCATCGAGTAAACCAGCTGACAAACAAGCCACAGAGAAGAAGAGCAA CTGGAAGGGTTAAGGGTGAAGACTCATCGAGTAAACCAGCTGACAAACAAGCCACAGAGAAGAAGAGCAA CTGGGAGAGTAAAAGGTGAAGATTCTTCAAATACGTCAGCTGATAAACAAGCCACAGATAAGAAGAACAA CTGGGAGAGTAAAAGGTGAAGATTCTTCAAATACGTCAGCTGATAAACAAGCCACAGATAAGAAGAACAA CTGGGAGAGTAAAAGGTGAAGATTCTTCAAATAAGTCAGCTGATAAACAAGCCACAGATAAGAAGAACAA GGTAAGTGAACAAGCTCAGCCACAATCTCGTCAGAGCGAAATGGAAGTACCTCAAGTTCGAGACAGAGAT GGTAAGTGGACAAGCTCAACCACAGTCTCGTCAGAGTGAAATGGAAGTACCCCAAGTTCGAGACAGAGAT GGTAAGTGGACAAGCTCAGCCACAATCTCGTCAGAGTGAAATGGAGGTACCCCAAGTTCGAGACAGAGAC GGTAAGTGGACAAGTTCAGCCACAATCTCGTCAGAGCGAAATGGAAGTACCTCAAGTTCGAGACAGAGAT GGTAAGTGGACAAGTTCAGCCACAATCTCGTCAGAGCGAAATGGAAGTACCTCAAGTTCGAGACAGAGAT GGTAAGTGGACAAGTTCAGCCACAATCTCGTCAGAGCGAAATGGAAGTACCTCAAGTTCGAGACAGAGAT Gambar 7.2 Perbandingan hasil sikuen nukleotida 5 isolat lemah ChiVMV dan isolat kuat CKB-2 (GeneBank aksesi DQ854960). Huruf dengan latar belakang hitam menunjukkan kesamaan sikuen, sedangkan huruf dengan latar belakang putih menunjukkan ketidaksamaan sikuen dengan isolat lainnya. Allignment menggunakan software Bioedit versi (Isis Pharmaceuticals, Inc).
7 GTTAATGTTGGGACCTCAGGGACATTCACAATACCGCGTCTCAAGGGTATTTCCTCAAAGTTAACAATAC GTTAATGTTGGGACCTCAGGGACATTCACAATACCGCGTCTCAAGGGTATTTCTTCAAAGTTAACAATAC GTTAATGTTGGGACCTCAGGGACATTCACAATACCGCGTCTCAAGGGTATTTCTTCAAAGTTAACAATAC GTTAATGTTGGGACCTCAGGGACATTCACAATACCGCGTCTTAAGGGTATTTCTTCAAAGTTAACAATAC GTTAATGTTGGGACCTCAGGGACATTCACAATACCGCGTCTTAAGGGTATTTCTTCAAAGTTAACAATAC GTTAATGTTGGGACCTCAGGGACATTCACAATACCGCGTCTTAAGGGTATTTCTTCAAAGTTAACAATAC CAAAGATTAAAACAAAGGCTGTTGTTAACCTTGAACACCTTCTTGATTATGCCCCTGAGCAAATACATTT CAAAGATCAAAACAAAGGCTGTTGTTAACCTTGAACACCTTCTTGATTATGCCCCTGAGCAAATACATTT CAAAGATCAAAACAAAGGCTGTTGTTAACCTTGAACACCTTCTTGATTATGCCCCTGAGCAAATACATTT CAAAGATTAAAACAAAGGCTGTTGTTAACCTTGAACACCTTCTTGATTATGCCCCTGAGCAAATACATTT CAAAGATTAAAACAAAGGCTGTTGTTAACCTTGAACACCTTCTTGATTATGCCCCTGAGCAAATACATTT CAAAGATTAAAACAAAGGCTGTTGTTAACCTTGAACACCTTCTTGATTATGCCCCTGAGCAAATACATTT GAGTAATACAAGGGCATTACAATCACAATTTGCATCCTGGTATGAAGGTGTTAAGAATGACTATGATGTC AAGTAATACAAGGGCATTACAATCACAATTTGCATCTTGGTATGAAGGTGTTAAGAATGACTATGATGTC AAGTAATACAAGGGCATTACAATCACAATTTGCATCTTGGTATGAAGGTGTTAAGAATGACTATGATGTC GAGTAATACAAGGGCATTACAATCACAATTTGCATCCTGGTATGAAGGTGTTAAGAATGACTACGATGTC GAGTAATACAAGGGCATTACAATCACAATTTGCATCCTGGTATGAAGGTGTTAAGAATGACTACGATGTC GAGTAATACAAGGGCATTACAATCACAATTTGCATCCTGGTATGAAGGTGTTAAGAATGACTATGATGTC ACAGATGAACAGATGCAAATAATACTGAATGGATTGATGGTTTGGTGTATTGAGAATGGAACGTCACCAA ACAGATGAACAGATGCAAATAATATTGAATGGATTGATGGTTTGGTGTATTGAGAATGGAACGTCACCAA ACAGATGAACAGATGCAAATAATATTGAATGGATTGATGGTTTGGTGTATTGAGAATGGAACGTCACCAA ACAGATGAACAGATGCAAATAATACTGAACGGATTGATGGTTTGGTGTATTGAGAATGGAACGTCACCAA ACAGATGAACAGATGCAAATAATACTGAACGGATTGATGGTTTGGTGTATTGAGAATGGAACGTCACCAA ACAGATGAACAGATGCAAATAATACTGAACGGATTGATGGTTTGGTGTATTGAGAATGGAGCGTCACCAA ACATCAATGGTTATTGGGTTATGATGGATGGAGATGAGCAAGTTGAATATCCAATAAAAGCCATTGATTG ATATCAACGGTTATTGGGTTATGATGGATGGAGATGAACAAGTTGAATATCCAATAAA-GCCATTGATTG ATATCAATGGTTTTTGGGTTATGATGGATGGAGATGAACAAGTTGAATATCCAATAAA-GCCATTGATTG ACATCAATGGTTATTGGGTTATGATGGATGGAGATGAACAAGTTGAATATCCAATAAA-GCCATTAATTG ACATCAATGGTTATTGGGTTATGATGGATGGAGATGAACAAGTTGAATATCCAATAAA-GCCATTAATTG ACATCAATGGTTATTGGGTTATGATGGATGGAGATGAACAAGTTGAATATCCAATAAA-GCCATTGATTG ACCATGCCAAACCATCATTTAGACAAATCATGGCGCATTTTAGCAATCTTGCTG-GAAAGCGTA ATCATGCTAAACCATCATTTAGACAAATCATGGCGCATTTTAGCAATCTTGCTGGAAGCGTAAA ATCATGCTAAACCTTCATTTAGACAAATCATGGCGCATTTTAGCAATCTTGCTGAAAGCGTAAA ACCATGCCAAACCATCATTTTGACAAATCATGGCGCATTTTAGCAATCTTGCTG-AAGCGTAAA ACCATGCCAAACCATCATTTACACAAATCATGGCGCATTTTAGCAATCTTGCTG-AAGCGTAAA ACCATGCCAAACCATCATTTAGACAAATCATGGCGCATTTTAGCAATCTTGCTG-AAGCGTATA Gambar 7.2.Lanjutan.
8 62 Berdasarkan hasil penyejajaran (allignment) sikuen nukleotida tersebut ditemukan perbedaan sebanyak 99 posisi basa yang tidak sama urutannya pada kelima ChiVMV isolat lemah dan satu isolat kuat ChiVMV-CKB. Terdapat 51 posisi basa yang sama pada isolat lemah ChiVMV- dan tetapi berbeda pada isolat lemah ChiVMV-,, dan. Ditemukan juga 5 posisi basa yang sama pada isolat lemah ChiVMV- dan tetapi berbeda pada isolat lemah ChiVMV-, dan (Gambar 7.2). Perbedaan sikuen antara isolat lemah dilaporkan pula oleh Wang et al. (2004) dimana isolat lemah ZYMV memiliki perbedaan sebanyak 14 nukleotida dengan isolat ZYMV Z5-1, walaupun kedua isolat mempunyai 9593 nukleotida yang mengkode poliprotein 3080 asam amino. Analisis Homologi Isolat-isolat Lemah ChiVMV Berdasarkan Sikuen nukleotida Gen CP Berdasarkan analisis homologi sikuen nukleotida gen CP kelima ChiVMV isolat lemah memiliki tingkat kesamaan satu sama lain 92.8% sampai 99.6%. Isolat lemah ChiVMV- dan memiliki hubungan paling dekat dengan tingkat kesamaan sikuen nukleotida 99.6%, dan isolat memiliki hubungan dekat dengan isolat lemah ChiVMV- dengan tingkat kesamaan sikuen nukleotida 97.5%. Kelima ChiVMV isolat lemah memiliki tingkat kesamaan berkisar 92.6% sampai 98.1% dengan isolat kuat ChiVMV-CKB (Tabel 7.2). Tingkat kesamaan yang tinggi antara isolat-isolat ChiVMV tersebut memberikan indikasi bahwa isolat ChiVMV Indonesia memiliki variasi genetik yang rendah. Menurut kriteria Moury et al. (2005) dan Zhao et al. (2003) bahkan dinyatakan bahwa isolat-isolat virus yang memiliki tingkat kesamaan 90% dapat dinyatakan sebagai isolat virus yang identik. Tingkat kesamaan yang tinggi juga terjadi antara kelima ChiVMV isolat lemah dengan ChiVMV isolat Pataruman yang diidentifikasi oleh Tsai et al. (2008) yaitu berkisar antara 92.8% sampai 99.6%, dengan isolat ChiVMV-S7 (93% sampai 94.1%), isolat ChiVMV-HCH9 (92% sampai 93.3%), dan isolat ChiVMV-D10 (91.8% sampai 93.5%) (Tabel 7.2).
9 63 Tabel 7.2 Homologi Chili veinal mottle virus isolat lemah dan isolat ChiVMV dari beberapa negara berdasarkan sikuen nukleotida gen CP No. Isolat Tingkat kesamaan (%)* CKB Pataruman Wenchang S HCH D Ca CM VN/C CTV_ * Tingkat kesamaan nukleotida dihitung menggunakan software Bioedit versi (Isis Pharmaceuticals, Inc). Gambar 7.3 Pohon filogenetika berdasarkan sikuen nukleotida Chili veinal mottle virus, dibuat dengan software Geenbee yang tersedia pada
10 64 Dari hasil allignment oleh Moury et al. (2005) antara sesama isolat ChiVMV mempunyai kesamaan sikuen nukleotida sebesar 90.3% sampai 92.8%, kemudian Tsai et al. (2008) menyatakan dari 24 isolat ChiVMV yang diuji ternyata menunjukkan kesamaan sikuen nukleotida sebesar 89.9% sampai 99.9%, dan dengan isolat ChiVMV meliputi PVBV, CVbMV dan CVbMV-CM1 sebagai pembanding menunjukkan kesamaan sikuen nukleotida sebesar 89.5% sampai 97.8%. Analisis filogenetik menggunakan perbedaan sikuen nukleotida dari 5 isolat ChiVMV yang diuji dan sikuen ChiVMV lain yang tersedia di GeneBank menunjukkan terbentuknya 2 kelompok, kelompok pertama terdiri dari isolat ChiVMV-,,, CKB, Pataruman,,, S7, HCH9, D10, dan VN/C2, kelompok kedua terdiri dari isolat ChiVMV-Ca, Wenchang, dan CM1. Kelima ChiVMV isolat lemah membentuk kelompok yang sama dengan isolat kuat ChiVMV-CKB, pengelompokan ini menunjukkan bahwa keenam isolat tersebut memiliki kekerabatan yang sangat erat. ChiVMV isolat lemah terbagi lagi menjadi 2 sub kelompok, yaitu sub kelompok pertama terdiri dari isolat lemah ChiVMV-,, dan, dan sub kelompok kedua yaitu isolat lemah ChiVMV- dan. Isolat lemah ChiVMV- dan dekat dengan isolat kuat ChiVMV-CKB, ke 3 isolat tersebut berasal dari pertanaman cabai di provinsi Jawa Barat. isolat lemah ChiVMV- lebih dekat dengan isolat lemah ChiVMV-, kedua isolat tersebut diperoleh dari provinsi Sumatera Barat. Sedangkan isolat lemah ChiVMV- yang diperoleh dari provinsi Jambi lebih dekat hubungan kekerabatannya dengan isolat ChiVMV dari Jawa Barat (Gambar 7.3). Hasil tersebut menunjukkan kecendrungan adanya hubungan kekerabatan yang berkorelasi dengan daerah asal isolat. Analisis Homologi Isolat-isolat Lemah ChiVMV Berdasarkan Sikuen Asam Amino Gen CP Berdasarkan hasil penyejajaran sikuen asam amino terdapat perbedaan sebanyak 51 posisi asam amino yang tidak sama pada kelima ChiVMV isolat lemah. Antara isolat lemah ChiVMV- dan terdapat perbedaan sebanyak 9 asam amino dengan isolat lemah ChiVMV-,, dan, sedangkan antara isolat lemah ChiVMV- dan dengan isolat lemah ChiVMV-, dan terdapat perbedaan sebanyak 2 asam amino (Gambar 7.4). Perbandingan sikuen asam amino antara 5 ChiVMV isolat lemah dengan 9 isolat ChiVMV dari berbagai negara menunjukkan kesamaan sikuen asam amino sebesar 88.0% sampai 99.6%. Perbandingan 5 isolat lemah ChiVMV dengan 2 isolat ChiVMV Indonesia (CKB dan Pataruman) menunjukkan kesamaan sikuen asam amino sebesar 90.8% sampai 97.5%. Perbandingan antara sesama ChiVMV isolat lemah menunjukkan kesamaan sikuen asam amino sebesar 90.5% sampai 99.6%. Penyejajaran antar sesama ChiVMV isolat lemah menunjukkan kesamaan sikuen asam amino tertinggi yaitu 99.6% antara isolat ChiVMV- dan (Tabel 7.3).
11 GV-FQF-TEGKTVQPEHRLEAICASMIEAWGYKELLHEIRLFYKWVIEQAPYSQLVSEGKAPYISETALR KRIVSILEWDRAEQPEHRLEAICASMIEAWGYKELLHEIRLFYKWVIEQAPYSQLVSEGKAPYISETALR RRIVSILEWDRAEQPEHRLEAICASMIEAWGYKELLHEIRLFYKWVIEQAPYSQLVSEGKAPYISETALR RRIVSILEWDRAEQPEHRLEAICASMIEAWGYRELLHEIRLFYKWVIEQAPYSQLVSEGKAPYISETALR KRIVSILEWDRAEKPEHRLEAICASMIEAWGYRELLHEIRLFYKWVIEQAPYSQLVSEGKAPYISETALR ERIVSILEWDRAEQPEHRLEAICASMIEAWGYKELLHEIRLFYKWVIEQAPYSQLVSEGKAPYISETALR CLYMSGHGESDISPYLRALIEGARREELDDESGEVTHQSGESVDAGRIKGEDSSNKSADKQATDKKSKVS CLYMSEHGESDISPYLRALIEGAKEEGLDDEGGEVAHQSGESVDAGRVKGEDSSSKPADKQATEKKSKVS CLYMSEHGESDISPYLRALIEGAKEEGLDDEGGEVAHQSGESIDAGRVKGEDSSSKPADKQATEKKSKVS CLYMSEHGESDISPYLRALIEGARREELNDEGGEVTHQSGESVDAGRVKGEDSSNTSADKQATDKKNKVS CLYMSEHGESDISPYLRALIEGARREELNDEGGEVTHQSGESVDAGRVKGEDSSNTSADKQATDKKNKVS CLYVSEHGESDISPYLRALIEGARREELNDEGGEVTHQSGESVDAGRVKGEDSSNKSADKQATDKKNKVS EQAQPQSRQSEMEVPQVRDRDVNVGTSGTFTIPRLKGISSKLTIPKIKTKAVVNLEHLLDYAPEQIHLSN GQAQPQSRQSEMEVPQVRDRDVNVGTSGTFTIPRLKGISSKLTIPKIKTKAVVNLEHLLDYAPEQIHLSN GQAQPQSRQSEMEVPQVRDRDVNVGTSGTFTIPRLKGISSKLTIPKIKTKAVVNLEHLLDYAPEQIHLSN GQVQPQSRQSEMEVPQVRDRDVNVGTSGTFTIPRLKGISSKLTIPKIKTKAVVNLEHLLDYAPEQIHLSN GQVQPQSRQSEMEVPQVRDRDVNVGTSGTFTIPRLKGISSKLTIPKIKTKAVVNLEHLLDYAPEQIHLSN GQVQPQSRQSEMEVPQVRDRDVNVGTSGTFTIPRLKGISSKLTIPKIKTKAVVNLEHLLDYAPEQIHLSN TRALQSQFASWYEGVKNDYDVTDEQMQIILNGLMVWCIENGTSPNINGYWVMMDGDEQVEYPIKAID-PC TRALQSQFASWYEGVKNDYDVTDEQMQIILNGLMVWCIENGTSPNINGYWVMMDGDEQVEYPIKPLIDHA TRALQSQFASWYEGVKNDYDVTDEQMQIILNGLMVWCIENGTSPNINGFWVMMDGDEQVEYPIKPLIDHA TRALQSQFASWYEGVKNDYDVTDEQMQIILNGLMVWCIENGTSPNINGYWVMMDGDEQVEYPIKPLIDHA TRALQSQFASWYEGVKNDYDVTDEQMQIILNGLMVWCIENGTSPNINGYWVMMDGDEQVEYPIKPLIDHA TRALQSQFASWYEGVKNDYDVTDEQMQIILNGLMVWCIENGASPNINGYWVMMDGDEQVEYPIKPLIDHA QTII-TNHGAFQSCWKA KPSFRQIMAHFSNLAGS KPSFRQIMAHFSNLAES KPSF-QIMAHFSNLAEA KPSFTQIMAHFSNLAEA KPSFRQIMAHFSNLAEA Gambar 7. 4 Perbandingan hasil sikuen asam amino 5 isolat lemah Chili veinal mottle virus dan isolat kuat CKB-2 (GeneBank aksesi DQ854960). Huruf dengan latar belakang hitam menunjukkan kesamaan sikuen, sedangkan huruf dengan latar belakang putih menunjukkan ketidaksamaan sikuen dengan isolat lainnya. Allignment menggunakan software Bioedit versi (Isis Pharmaceuticals, Inc).
12 66 Tabel 7.3 Homologi Chili veinal mottle virus isolat lemah dan isolat ChiVMV dari beberapa negara berdasarkan sikuen asam amino gen CP No Isolat Tingkat kesamaan (%)* CKB Pataruman Wenchang S HCH D Ca CM VN/C CTV_ * Tingkat kesamaan asam amino dihitung menggunakan software Bioedit versi (Isis Pharmaceuticals, Inc). Gambar 7.5 Pohon filogenetika berdasarkan sikuen asam amino Chili veinal mottle virus, dibuat dengan software Geenbee yang tersedia pada
13 67 Antara 5 ChiVMV isolat lemah (,,,, dan ) dengan isolat kuat ChiVMV-CKB menunjukkan kesamaan sikuen asam amino masingmasing berturut-turut 90.8%, 93.2%, 92.3%, 97.5%, dan 97.5%. Kesamaan sikuen asam amino antara ChiVMV isolat lemah dengan isolat kuat ChiVMV-CKB tertinggi yaitu 97.5% terdapat pada isolat lemah ChiVMV- dan, sedangkan kesamaan sikuen asam amino terendah (90.8%) terdapat pada isolat lemah ChiVMV-. Walaupun demikian secara umum kesamaan sikuen asam amino kelima ChiVMV isolat lemah dengan isolat kuat ChiVMV-CKB tergolong tinggi yaitu 90%. Perbandingan sikuen asam amino antara 7 isolat ChiVMV Indonesia (5 ChiVMV isolat lemah, CKB, dan Pataruman) dengan isolat ChiVMV dari negara lain menunjukkan kesamaan tertinggi (90.9% sampai 98.1%) dengan isolat D10 dari India, dan kesamaan terendah (87.2% sampai 94.9%) dengan isolat VN/C2 dari Vietnam (Tabel 7.3). Analisis kekerabatan sesama isolat ChiVMV yang digambarkan melalui pohon filogenetika asam amino menghasilkan 2 kelompok. Kelompok pertama terdiri dari isolat,,, CKB, Pataruman,,, S7, HCH9, D10, CM1, Wenchang, Ca, kelompok kedua adalah isolat VN/C2. ChiVMV isolat lemah terpecah menjadi 2 sub kelompok sama halnya dengan filogenetika nukleotida, sub kelompok pertama adalah ChiVMV isolat lemah (,, dan ), dan sub kelompok kedua adalah ChiVMV isolat lemah (, dan ). Bila dibandingkan antara pohon filogenetika nukleotida dengan asam amino tidak terdapat perbedaan pengelompokan ChiVMV isolat lemah, dengan demikian dapat dikatakan mutasi nukleotida pada ChiVMV isolat lemah tidak bermakna terhadap pohon filogenetika asam amino (Gambar 7.5). Pentingnya Karakter Molekuler Isolat Virus untuk Penerapan Proteksi Silang Derajat kesamaan sikuen nukleotida dan asam amino antara ChiVMV isolat lemah dengan isolat kuat ChiVMV-CKB dapat menentukan efektivitas proteksi silang. Dilaporkan oleh Hanssen et al. (2010) bahwa proteksi silang pada tanaman tomat yang terinfeksi Pepino mosaic virus (PepMV) lebih baik hasilnya bila menggunakan isolat lemah PepMV-CH2 dibandingkan menggunakan isolat lemah PepMV-EU atau -LP. Ternyata kesamaan sikuen nukleotida isolat lemah PepMV-CH2 dengan isolat kuat PepMV adalah 99.4%, sedangkan antara isolat lemah PepMV-EU dan -LP dengan isolat kuat adalah 79%. Hal yang sama dilaporkan untuk kasus Papaya ringspot virus (PRSV) oleh Yeh dan Gonsalves (1984) yaitu isolat lemah PRSV-P lebih efisien dibandingkan PRSV-W. Albiach-Marti et al. (2000) menyatakan bahwa CTV strain lemah mampu melindungi tanaman jeruk di Florida dan Spanyol karena strain lemah tersebut memiliki kesamaan sikuen yang tinggi terhadap CTV strain kuat. Lebih lanjut Zhou et al. (2002) membuktikan bahwa isolat lemah CTV-PB61 lebih efektif melindungi tanaman jeruk terhadap isolat kuat CTV-PB 235 dari pada isolat CTV-PB 155. Kesamaan sikuen nukleotida CP antara CTV-PB61 dan CTV-PB 235 lebih tinggi dari pada antara CTV-PB61 dan CTV-PB 155 yaitu masingmasing 97.6% dan 91.2%. Proteksi silang berhasil bila tanaman tidak menimbulkan gejala atau hanya menimbulkan gejala lemah setelah inokulasi strain kuat, sedangkan pada kontrol
14 68 (inokulasi dengan strain kuat saja) memperlihatkan gejala yang parah (You et al. 2005). Gen CP berperan penting dalam proteksi silang, karena coat protein virus strain lemah akan menghalangi uncoating virus strain kuat yang menginfeksi setelah strain lemah (Culver 1996; You et al. 2005). Selain mekanisme menghalangi uncoating strain kuat, mekanisme proteksi silang kemungkinan berkaitan dengan induksi RNA silencing (Gal-on dan Shiboleth 2006). Kedua mekanisme tersebut berlangsung dengan efisien bila virus-virus yang menginfeksi tanaman inang yang sama memiliki hubungan kekerabatan yang dekat. KESIMPULAN Karakter molekuler ChiVMV isolat lemah (,,,, dan ) menunjukkan kesamaan sikuen nukleotida dan asam amino yang lebih dekat dengan isolat ChiVMV dari Indonesia (isolat Pataruman) dan India (isolat S7). Analisis filogenetika sikuen nukleotida dan asam amino menunjukkan bahwa kelima ChiVMV isolat lemah berada pada kelompok yang sama dengan isolat kuat ChiVMV-CKB.
BAHAN DAN METODE Tempat dan Waktu Penelitian Bahan dan Alat
 12 BAHAN DAN METODE Tempat dan Waktu Penelitian Survei penyakit klorosis dan koleksi sampel tanaman tomat sakit dilakukan di sentra produksi tomat di daerah Cianjur, Cipanas, Lembang, dan Garut. Deteksi
12 BAHAN DAN METODE Tempat dan Waktu Penelitian Survei penyakit klorosis dan koleksi sampel tanaman tomat sakit dilakukan di sentra produksi tomat di daerah Cianjur, Cipanas, Lembang, dan Garut. Deteksi
BAHAN DAN METODE. Metode Penelitian
 BAHAN DAN METODE Tempat dan Waktu Penelitian Survei penyakit mosaik dan koleksi sampel tanaman nilam sakit dilakukan di Kebun Percobaan Balai Tanaman Obat dan Aromatik (BALITTRO) di daerah Gunung Bunder
BAHAN DAN METODE Tempat dan Waktu Penelitian Survei penyakit mosaik dan koleksi sampel tanaman nilam sakit dilakukan di Kebun Percobaan Balai Tanaman Obat dan Aromatik (BALITTRO) di daerah Gunung Bunder
ISOLAT LEMAH SEBAGAI AGENS PROTEKSI SILANG PADA TANAMAN CABAI
 41 6 EVLUSI KEMMPUN Chili veinal mottle virus ISOLT LEMH SEGI GENS PROTEKSI SILNG PD TNMN CI (Evaluation of Weak Isolates of Chili veinal mottle virus for gent of Cross Protection in Chili Pepper) bstrak
41 6 EVLUSI KEMMPUN Chili veinal mottle virus ISOLT LEMH SEGI GENS PROTEKSI SILNG PD TNMN CI (Evaluation of Weak Isolates of Chili veinal mottle virus for gent of Cross Protection in Chili Pepper) bstrak
HASIL DAN PEMBAHASAN
 HASIL DAN PEMBAHASAN Deteksi Fabavirus pada Tanaman Nilam Deteksi Fabavirus Melalui Uji Serologi Tanaman nilam dari sampel yang telah dikoleksi dari daerah Cicurug dan Gunung Bunder telah berhasil diuji
HASIL DAN PEMBAHASAN Deteksi Fabavirus pada Tanaman Nilam Deteksi Fabavirus Melalui Uji Serologi Tanaman nilam dari sampel yang telah dikoleksi dari daerah Cicurug dan Gunung Bunder telah berhasil diuji
Abstrak. Kata kunci : Chili veinal mottle virus, isolat lemah, isolat kuat. Abstract
 31 5 INTERAKSI ANTARA Chili veinal mottle virus ISOLAT LEMAH DENGAN ISOLAT KUAT (Interaction between Weak Isolates and Severe Isolate of Chili veinal mottle virus) Abstrak Salah satu virus yang banyak
31 5 INTERAKSI ANTARA Chili veinal mottle virus ISOLAT LEMAH DENGAN ISOLAT KUAT (Interaction between Weak Isolates and Severe Isolate of Chili veinal mottle virus) Abstrak Salah satu virus yang banyak
IDENTIFIKASI SPESIES POTYVIRUS PENYEBAB PENYAKIT MOSAIK PADA TANAMAN CABAI (Capsicum frutescens L.) MELALUI SIKUEN NUKLEOTIDA GEN Coat Protein
 IDENTIFIKASI SPESIES POTYVIRUS PENYEBAB PENYAKIT MOSAIK PADA TANAMAN CABAI (Capsicum frutescens L.) MELALUI SIKUEN NUKLEOTIDA GEN Coat Protein I Gede Rian Pramarta 1, I Gede Rai Maya Temaja 1*), I Dewa
IDENTIFIKASI SPESIES POTYVIRUS PENYEBAB PENYAKIT MOSAIK PADA TANAMAN CABAI (Capsicum frutescens L.) MELALUI SIKUEN NUKLEOTIDA GEN Coat Protein I Gede Rian Pramarta 1, I Gede Rai Maya Temaja 1*), I Dewa
HASIL DAN PEMBAHASAN
 HASIL DAN PEMBAHASAN Kisaran Inang Potyvirus Isolat Nilam Bogor Tanaman nilam sakit banyak terdapat di daerah Bogor yang memperlihatkan gejala mosaik dengan ciri-ciri hampir sama dengan yang pernah diutarakan
HASIL DAN PEMBAHASAN Kisaran Inang Potyvirus Isolat Nilam Bogor Tanaman nilam sakit banyak terdapat di daerah Bogor yang memperlihatkan gejala mosaik dengan ciri-ciri hampir sama dengan yang pernah diutarakan
MATERI DAN METODE. Kota Padang Sumatera Barat pada bulan Oktober Amplifikasi gen Growth
 III. MATERI DAN METODE 3.1 Waktu dan Tempat Pengambilan sampel darah domba dilakukan di Kecamatan Koto Tengah Kota Padang Sumatera Barat pada bulan Oktober 2012. Amplifikasi gen Growth Hormone menggunakan
III. MATERI DAN METODE 3.1 Waktu dan Tempat Pengambilan sampel darah domba dilakukan di Kecamatan Koto Tengah Kota Padang Sumatera Barat pada bulan Oktober 2012. Amplifikasi gen Growth Hormone menggunakan
BAHAN DAN METODE Tempat dan Waktu Metode Penelitian Pengambilan Sampel Kutukebul dan Tanaman Tomat Sumber TICV
 BAHAN DAN METODE Tempat dan Waktu Kegiatan survei dan pengambilan sampel kutukebul dilakukan di sentra produksi tomat di Kecamatan Cikajang (kabupaten Garut), Kecamatan Pacet (Kabupaten Cianjur), Kecamatan
BAHAN DAN METODE Tempat dan Waktu Kegiatan survei dan pengambilan sampel kutukebul dilakukan di sentra produksi tomat di Kecamatan Cikajang (kabupaten Garut), Kecamatan Pacet (Kabupaten Cianjur), Kecamatan
BAB III. METODOLOGI PENELITIAN. Pengambilan sampel. Penyiapan templat mtdna dengan metode lisis sel
 16 BAB III. METODOLOGI PENELITIAN Bab ini menggambarkan tahapan penelitian yang terdiri dari pengambilan sampel, penyiapan templat mtdna dengan metode lisis sel, amplifikasi D-loop mtdna dengan teknik
16 BAB III. METODOLOGI PENELITIAN Bab ini menggambarkan tahapan penelitian yang terdiri dari pengambilan sampel, penyiapan templat mtdna dengan metode lisis sel, amplifikasi D-loop mtdna dengan teknik
MATERI DAN METODE. Lokasi dan Waktu. Materi. Tabel 1. Jumah Sampel Darah Ternak Sapi Indonesia Ternak n Asal Sapi Bali 2 4
 MATERI DAN METODE Lokasi dan Waktu Penelitian dilaksanakan di Laboratorium Genetika Molekuler Ternak, Bagian Pemuliaan dan Genetika Ternak, Fakultas Peternakan, Institut Pertanian Bogor. penelitian ini
MATERI DAN METODE Lokasi dan Waktu Penelitian dilaksanakan di Laboratorium Genetika Molekuler Ternak, Bagian Pemuliaan dan Genetika Ternak, Fakultas Peternakan, Institut Pertanian Bogor. penelitian ini
METODOLOGI PENELITIAN. Tempat dan Waktu Penelitian. Bahan dan Alat Penelitian
 14 METODOLOGI PENELITIAN Tempat dan Waktu Penelitian Tempat penelitian dilakukan di Laboratorium Unit Pelayanan Mikrobiologi Terpadu, Bagian Mikrobiologi Kesehatan, Departemen Ilmu Penyakit Hewan dan Kesehatan
14 METODOLOGI PENELITIAN Tempat dan Waktu Penelitian Tempat penelitian dilakukan di Laboratorium Unit Pelayanan Mikrobiologi Terpadu, Bagian Mikrobiologi Kesehatan, Departemen Ilmu Penyakit Hewan dan Kesehatan
PRAKATA. Alhamdulillah syukur senantiasa penulis panjatkan kepada Allah swt., atas
 PRAKATA Alhamdulillah syukur senantiasa penulis panjatkan kepada Allah swt., atas segala nikmat dan karunia-nya, penulisan Tugas Akhir dengan judul Keragaman Genetik Abalon (Haliotis asinina) Selat Lombok
PRAKATA Alhamdulillah syukur senantiasa penulis panjatkan kepada Allah swt., atas segala nikmat dan karunia-nya, penulisan Tugas Akhir dengan judul Keragaman Genetik Abalon (Haliotis asinina) Selat Lombok
BAB III METODOLOGI PENELITIAN. Penelitian akan diawali dengan preparasi alat dan bahan untuk sampling
 16 BAB III METODOLOGI PENELITIAN Penelitian akan diawali dengan preparasi alat dan bahan untuk sampling sel folikel akar rambut. Sampel kemudian dilisis, diamplifikasi dan disekuensing dengan metode dideoksi
16 BAB III METODOLOGI PENELITIAN Penelitian akan diawali dengan preparasi alat dan bahan untuk sampling sel folikel akar rambut. Sampel kemudian dilisis, diamplifikasi dan disekuensing dengan metode dideoksi
BAB III METODE PENELITIAN. Dalam penelitian ini dilakukan lima tahap utama yang meliputi tahap
 BAB III METODE PENELITIAN Dalam penelitian ini dilakukan lima tahap utama yang meliputi tahap penyiapan templat mtdna, amplifikasi fragmen mtdna pada daerah D-loop mtdna manusia dengan teknik PCR, deteksi
BAB III METODE PENELITIAN Dalam penelitian ini dilakukan lima tahap utama yang meliputi tahap penyiapan templat mtdna, amplifikasi fragmen mtdna pada daerah D-loop mtdna manusia dengan teknik PCR, deteksi
3 METODOLOGI 3.1 Waktu dan Tempat 3.2 Bahan dan Alat
 3 METODOLOGI 3.1 Waktu dan Tempat Penelitian Autentikasi Bahan Baku Ikan Tuna (Thunnus sp.) dalam Rangka Peningkatan Keamanan Pangan dengan Metode Berbasis DNA dilaksanakan pada bulan Januari sampai dengan
3 METODOLOGI 3.1 Waktu dan Tempat Penelitian Autentikasi Bahan Baku Ikan Tuna (Thunnus sp.) dalam Rangka Peningkatan Keamanan Pangan dengan Metode Berbasis DNA dilaksanakan pada bulan Januari sampai dengan
BAB III METODE PENELITIAN
 BAB III METODE PENELITIAN A. Jenis Penelitian deskriptif. Jenis penelitian yang digunakan adalah penelitian dasar dengan metode B. Objek Penelitian Objek penelitian ini adalah sampel DNA koleksi hasil
BAB III METODE PENELITIAN A. Jenis Penelitian deskriptif. Jenis penelitian yang digunakan adalah penelitian dasar dengan metode B. Objek Penelitian Objek penelitian ini adalah sampel DNA koleksi hasil
HASIL DAN PEMBAHASAN
 HASIL DAN PEMBAHASAN Virus Hepatitis B Gibbon Regio Pre-S1 Amplifikasi Virus Hepatitis B Regio Pre-S1 Hasil amplifikasi dari 9 sampel DNA owa jawa yang telah berstatus serologis positif terhadap antigen
HASIL DAN PEMBAHASAN Virus Hepatitis B Gibbon Regio Pre-S1 Amplifikasi Virus Hepatitis B Regio Pre-S1 Hasil amplifikasi dari 9 sampel DNA owa jawa yang telah berstatus serologis positif terhadap antigen
BAB III METODOLOGI PENELITIAN. Pada penelitian ini terdapat lima tahapan penelitian yang dilakukan yaitu
 BAB III METODOLOGI PENELITIAN Pada penelitian ini terdapat lima tahapan penelitian yang dilakukan yaitu pengumpulan sampel berupa akar rambut, ekstraksi mtdna melalui proses lisis akar rambut, amplifikasi
BAB III METODOLOGI PENELITIAN Pada penelitian ini terdapat lima tahapan penelitian yang dilakukan yaitu pengumpulan sampel berupa akar rambut, ekstraksi mtdna melalui proses lisis akar rambut, amplifikasi
FAKULTAS BIOLOGI LABORATORIUM GENETIKA & PEMULIAAN INSTRUKSI KERJA UJI
 ISOLASI TOTAL DNA TUMBUHAN DENGAN KIT EKSTRAKSI DNA PHYTOPURE Halaman : 1 dari 5 1. RUANG LINGKUP Metode ini digunakan untuk mengisolasi DNA dari sampel jaringan tumbuhan, dapat dari daun, akar, batang,
ISOLASI TOTAL DNA TUMBUHAN DENGAN KIT EKSTRAKSI DNA PHYTOPURE Halaman : 1 dari 5 1. RUANG LINGKUP Metode ini digunakan untuk mengisolasi DNA dari sampel jaringan tumbuhan, dapat dari daun, akar, batang,
BAB III METODE PENELITIAN
 39 BAB III METODE PENELITIAN A. Desain Penelitian Penelitian yang dilakukan merupakan penelitian deskriptif. Penelitian membuat deskripsi secara sistematis, faktual dan akurat mengenai fakta-fakta dan
39 BAB III METODE PENELITIAN A. Desain Penelitian Penelitian yang dilakukan merupakan penelitian deskriptif. Penelitian membuat deskripsi secara sistematis, faktual dan akurat mengenai fakta-fakta dan
4.1. Alat dan Bahan Penelitian a. Alat Penelitian. No. URAIAN ALAT. A. Pengambilan sampel
 7 IV. METODE PENELITIAN Ikan Lais diperoleh dari hasil penangkapan ikan oleh nelayan dari sungaisungai di Propinsi Riau yaitu S. Kampar dan S. Indragiri. Identifikasi jenis sampel dilakukan dengan menggunakan
7 IV. METODE PENELITIAN Ikan Lais diperoleh dari hasil penangkapan ikan oleh nelayan dari sungaisungai di Propinsi Riau yaitu S. Kampar dan S. Indragiri. Identifikasi jenis sampel dilakukan dengan menggunakan
BAB III BAHAN DAN METODE
 9 BAB III BAHAN DAN METODE 3.1 Waktu dan Tempat Penelitian Penelitian dilaksanakan pada bulan September 2011 sampai dengan Juli 2012. Kegiatan ekstraksi DNA sampai PCR-RFLP dilakukan di laboratorium Analisis
9 BAB III BAHAN DAN METODE 3.1 Waktu dan Tempat Penelitian Penelitian dilaksanakan pada bulan September 2011 sampai dengan Juli 2012. Kegiatan ekstraksi DNA sampai PCR-RFLP dilakukan di laboratorium Analisis
BAB III BAHAN DAN CARA KERJA. Penelitian dilakukan di Laboratorium Institute of Human Virology and
 23 BAB III BAHAN DAN CARA KERJA A. LOKASI DAN WAKTU PENELITIAN Penelitian dilakukan di Laboratorium Institute of Human Virology and Cancer Biology of the University of Indonesia (IHVCB-UI), Jl. Salemba
23 BAB III BAHAN DAN CARA KERJA A. LOKASI DAN WAKTU PENELITIAN Penelitian dilakukan di Laboratorium Institute of Human Virology and Cancer Biology of the University of Indonesia (IHVCB-UI), Jl. Salemba
BAHAN DAN METODE Waktu dan Tempat Bahan Metode Isolasi C. gloeosporioides dari Buah Avokad
 15 BAHAN DAN METODE Waktu dan Tempat Penelitian dilaksanakan di Laboratorium Balai Besar Karantina Pertanian (BBKP) Tanjung Priok Wilayah Kerja Bogor, mulai bulan Oktober 2011 sampai Februari 2012. Bahan
15 BAHAN DAN METODE Waktu dan Tempat Penelitian dilaksanakan di Laboratorium Balai Besar Karantina Pertanian (BBKP) Tanjung Priok Wilayah Kerja Bogor, mulai bulan Oktober 2011 sampai Februari 2012. Bahan
Asam Asetat Glacial = 5,7 ml EDTA 0,5 M ph 8.0 = 10 ml Aquades ditambahkan hingga volume larutan 100 ml
 36 Lampiran 1. Pembuatan Larutan Stok dan Buffer A. Pembuatan Larutan Stok Tris HCL 1 M ph 8.0 (100 ml) : Timbang Tris sebanyak 12,114 g. Masukkan Tris ke dalam Erlenmeyer dan ditambahkan 80 ml aquades.
36 Lampiran 1. Pembuatan Larutan Stok dan Buffer A. Pembuatan Larutan Stok Tris HCL 1 M ph 8.0 (100 ml) : Timbang Tris sebanyak 12,114 g. Masukkan Tris ke dalam Erlenmeyer dan ditambahkan 80 ml aquades.
III. BAHAN DAN METODE
 III. BAHAN DAN METODE Tempat dan Waktu Penelitian dilakukan di Laboratorium BIORIN (Biotechnology Research Indonesian - The Netherlands) Pusat Penelitian Sumberdaya Hayati dan Bioteknologi IPB. Penelitian
III. BAHAN DAN METODE Tempat dan Waktu Penelitian dilakukan di Laboratorium BIORIN (Biotechnology Research Indonesian - The Netherlands) Pusat Penelitian Sumberdaya Hayati dan Bioteknologi IPB. Penelitian
3 METODE. Tempat dan Waktu
 13 3 METODE Tempat dan Waktu Penelitian terdiri atas dua kegiatan utama, yaitu (1) survei kejadian penyakit di lapangan dan (2) deteksi virus dan identifikasi kutukebul. Kegiatan pertama dilakukan di areal
13 3 METODE Tempat dan Waktu Penelitian terdiri atas dua kegiatan utama, yaitu (1) survei kejadian penyakit di lapangan dan (2) deteksi virus dan identifikasi kutukebul. Kegiatan pertama dilakukan di areal
BAB III METODE PENELITIAN. amplifikasi daerah HVI mtdna sampel dengan menggunakan teknik PCR;
 BAB III METODE PENELITIAN Secara garis besar, langkah-langkah yang dilakukan dalam penelitian ini adalah: pengumpulan sampel; lisis terhadap sampel mtdna yang telah diperoleh; amplifikasi daerah HVI mtdna
BAB III METODE PENELITIAN Secara garis besar, langkah-langkah yang dilakukan dalam penelitian ini adalah: pengumpulan sampel; lisis terhadap sampel mtdna yang telah diperoleh; amplifikasi daerah HVI mtdna
BAHAN DAN METODE Waktu dan Tempat Bahan dan Alat
 12 BAHAN DAN METODE Waktu dan Tempat Penelitian ini dilakukan dari bulan April 2006 sampai dengan bulan April 2007. Penelitian dilakukan di rumah kaca, laboratorium Biologi Molekuler Seluler Tanaman, dan
12 BAHAN DAN METODE Waktu dan Tempat Penelitian ini dilakukan dari bulan April 2006 sampai dengan bulan April 2007. Penelitian dilakukan di rumah kaca, laboratorium Biologi Molekuler Seluler Tanaman, dan
BAB III METODE PENELITIAN
 BAB III METODE PENELITIAN A. Jenis Penelitian Jenis penelitian yang dilakukan adalah penelitian deskriptif. Penelitian deskriptif adalah penelitian yang mendeskripsikan suatu gambaran yang sistematis dengan
BAB III METODE PENELITIAN A. Jenis Penelitian Jenis penelitian yang dilakukan adalah penelitian deskriptif. Penelitian deskriptif adalah penelitian yang mendeskripsikan suatu gambaran yang sistematis dengan
BAB III METODE PENELITIAN. Penelitian ini merupakan penelitian eksperimental dengan 7 sampel dari 7
 BAB III METODE PENELITIAN 3.1 Rancangan Penelitian Penelitian ini merupakan penelitian eksperimental dengan 7 sampel dari 7 individu udang Jari yang diambil dari Segara Anakan Kabupaten Cilacap Jawa Tengah.
BAB III METODE PENELITIAN 3.1 Rancangan Penelitian Penelitian ini merupakan penelitian eksperimental dengan 7 sampel dari 7 individu udang Jari yang diambil dari Segara Anakan Kabupaten Cilacap Jawa Tengah.
II. MATERI DAN METODE. Tempat pengambilan sampel daun jati (Tectona grandis Linn. f.) dilakukan di
 II. MATERI DAN METODE 2.1 Waktu dan Tempat Penelitian Tempat pengambilan sampel daun jati (Tectona grandis Linn. f.) dilakukan di enam desa yaitu tiga desa di Kecamatan Grokgak dan tiga desa di Kecamatan
II. MATERI DAN METODE 2.1 Waktu dan Tempat Penelitian Tempat pengambilan sampel daun jati (Tectona grandis Linn. f.) dilakukan di enam desa yaitu tiga desa di Kecamatan Grokgak dan tiga desa di Kecamatan
BAB III METODE PENELITIAN Bagan Alir Penelitian ini secara umum dapat digambarkan pada skema berikut:
 BAB III METODE PENELITIAN Tahapan-tahapan yang dilakukan dalam penelitian ini adalah: pengumpulan sampel, lisis terhadap sampel mtdna yang telah diperoleh, amplifikasi daerah HVI mtdna sampel dengan menggunakan
BAB III METODE PENELITIAN Tahapan-tahapan yang dilakukan dalam penelitian ini adalah: pengumpulan sampel, lisis terhadap sampel mtdna yang telah diperoleh, amplifikasi daerah HVI mtdna sampel dengan menggunakan
BAB III METODE PENELITIAN. Secara garis besar langkah-langkah yang dilakukan dalam penelitian ini
 BAB III METODE PENELITIAN Secara garis besar langkah-langkah yang dilakukan dalam penelitian ini adalah: pengumpulan sampel; lisis terhadap sampel mtdna yang telah diperoleh; amplifikasi daerah D-loop
BAB III METODE PENELITIAN Secara garis besar langkah-langkah yang dilakukan dalam penelitian ini adalah: pengumpulan sampel; lisis terhadap sampel mtdna yang telah diperoleh; amplifikasi daerah D-loop
BAB III METODE PENELITIAN
 BAB III METODE PENELITIAN A. Jenis penelitian Jenis penelitian ini adalah penelitian deskriptif yang mengangkat fenomena alam sebagai salah satu masalah dalam penelitian, sehingga dapat menerangkan arti
BAB III METODE PENELITIAN A. Jenis penelitian Jenis penelitian ini adalah penelitian deskriptif yang mengangkat fenomena alam sebagai salah satu masalah dalam penelitian, sehingga dapat menerangkan arti
BAB 4. METODE PENELITIAN
 BAB 4. METODE PENELITIAN Penelitian penanda genetik spesifik dilakukan terhadap jenis-jenis ikan endemik sungai paparan banjir Riau yaitu dari Genus Kryptopterus dan Ompok. Penelitian ini bertujuan untuk
BAB 4. METODE PENELITIAN Penelitian penanda genetik spesifik dilakukan terhadap jenis-jenis ikan endemik sungai paparan banjir Riau yaitu dari Genus Kryptopterus dan Ompok. Penelitian ini bertujuan untuk
3. METODE PENELITIAN
 19 3. METODE PENELITIAN 3.1 Waktu dan Tempat Penelitian ini dilaksanakan pada bulan Maret sampai Juni 2010 di Laboratorium Mikrobiologi, Biokimia dan Bioteknologi Hasil Perairan Departemen Teknologi Hasil
19 3. METODE PENELITIAN 3.1 Waktu dan Tempat Penelitian ini dilaksanakan pada bulan Maret sampai Juni 2010 di Laboratorium Mikrobiologi, Biokimia dan Bioteknologi Hasil Perairan Departemen Teknologi Hasil
LAMPIRAN. Lampiran 1. Pembuatan Larutan Stok dan Buffer
 LAMPIRAN Lampiran 1. Pembuatan Larutan Stok dan Buffer A. LARUTAN STOK CTAB 5 % (100 ml) - Ditimbang NaCl sebanyak 2.0 gram - Ditimbang CTAB sebanyak 5.0 gram. - Dimasukkan bahan kimia ke dalam erlenmeyer
LAMPIRAN Lampiran 1. Pembuatan Larutan Stok dan Buffer A. LARUTAN STOK CTAB 5 % (100 ml) - Ditimbang NaCl sebanyak 2.0 gram - Ditimbang CTAB sebanyak 5.0 gram. - Dimasukkan bahan kimia ke dalam erlenmeyer
LAMPIRAN. Lampiran 1. Deskripsi Pembuatan Larutan Stok dan Buffer
 LAMPIRAN Lampiran 1. Deskripsi Pembuatan Larutan Stok dan Buffer 1. Pembuatan Larutan Stok a. CTAB 5 % Larutan dibuat dengan melarutkan : - NaCl : 2.0 gr - CTAB : 5.0 gr - Aquades : 100 ml b. Tris HCl
LAMPIRAN Lampiran 1. Deskripsi Pembuatan Larutan Stok dan Buffer 1. Pembuatan Larutan Stok a. CTAB 5 % Larutan dibuat dengan melarutkan : - NaCl : 2.0 gr - CTAB : 5.0 gr - Aquades : 100 ml b. Tris HCl
BAB III METODE PENELITIAN
 BAB III METODE PENELITIAN A. Jenis Penelitian Jenis penelitian ini adalah penelitian deskriptif yang mengangkat fenomena alam sebagai salah satu masalah dalam penelitian. Penelitian ini dapat menerangkan
BAB III METODE PENELITIAN A. Jenis Penelitian Jenis penelitian ini adalah penelitian deskriptif yang mengangkat fenomena alam sebagai salah satu masalah dalam penelitian. Penelitian ini dapat menerangkan
Bab III Bahan dan Metode III.1 Bahan III. 2 Alat
 Bab III Bahan dan Metode III.1 Bahan Pada penelitian ini, sampel yang digunakan dalam penelitian, adalah cacing tanah spesies L. rubellus yang berasal dari peternakan cacing tanah lokal di Sekeloa, Bandung.
Bab III Bahan dan Metode III.1 Bahan Pada penelitian ini, sampel yang digunakan dalam penelitian, adalah cacing tanah spesies L. rubellus yang berasal dari peternakan cacing tanah lokal di Sekeloa, Bandung.
Identifikasi Carmovirus pada Tanaman Anyelir melalui Teknik Reverse Transcription-Polymerase Chain Reaction dan Analisis Sikuen Nukleotida
 ISSN:0215-7950 Volume 10, Nomor 3, Juni 2014 Halaman 87 92 DOI: 10.14692/jfi.10.3.87 Identifikasi Carmovirus pada Tanaman Anyelir melalui Teknik Reverse Transcription-Polymerase Chain Reaction dan Analisis
ISSN:0215-7950 Volume 10, Nomor 3, Juni 2014 Halaman 87 92 DOI: 10.14692/jfi.10.3.87 Identifikasi Carmovirus pada Tanaman Anyelir melalui Teknik Reverse Transcription-Polymerase Chain Reaction dan Analisis
BAB III METODE PENELITIAN
 BAB III METODE PENELITIAN A. Jenis Penelitian Jenis penelitian yang dilakukan merupakan penelitian deskriptif. Data diperoleh dari sikuen DNA daerah ITS untuk merekonstruksi pohon filogenetik dalam menentukan
BAB III METODE PENELITIAN A. Jenis Penelitian Jenis penelitian yang dilakukan merupakan penelitian deskriptif. Data diperoleh dari sikuen DNA daerah ITS untuk merekonstruksi pohon filogenetik dalam menentukan
IDENTIFIKASI MOLEKULER VIRUS PENYEBAB PENYAKIT DAUN KUNING PADA TANAMAN MENTIMUN DI KECAMATAN BATURITI KABUPATEN TABANAN
 TESIS IDENTIFIKASI MOLEKULER VIRUS PENYEBAB PENYAKIT DAUN KUNING PADA TANAMAN MENTIMUN DI KECAMATAN BATURITI KABUPATEN TABANAN I DEWA MADE PUTRA WIRATAMA PROGRAM PASCASARJANA UNIVERSITAS UDAYANA DENPASAR
TESIS IDENTIFIKASI MOLEKULER VIRUS PENYEBAB PENYAKIT DAUN KUNING PADA TANAMAN MENTIMUN DI KECAMATAN BATURITI KABUPATEN TABANAN I DEWA MADE PUTRA WIRATAMA PROGRAM PASCASARJANA UNIVERSITAS UDAYANA DENPASAR
HASIL DAN PEMBAHASAN TICV Isolat Indonesia
 23 HASIL DAN PEMBAHASAN TICV Isolat Indonesia Penyakit klorosis saat ini sudah ditemukan di Indonesia. Pertama kali ditemukan di sentra pertanaman tomat di Magelang, Jawa Tengah dan Purwakarta, Jawa Barat
23 HASIL DAN PEMBAHASAN TICV Isolat Indonesia Penyakit klorosis saat ini sudah ditemukan di Indonesia. Pertama kali ditemukan di sentra pertanaman tomat di Magelang, Jawa Tengah dan Purwakarta, Jawa Barat
4 Hasil dan Pembahasan
 4 Hasil dan Pembahasan Dalam bab ini akan dipaparkan hasil dari tahap-tahap penelitian yang telah dilakukan. Melalui tahapan tersebut diperoleh urutan nukleotida sampel yang positif diabetes dan sampel
4 Hasil dan Pembahasan Dalam bab ini akan dipaparkan hasil dari tahap-tahap penelitian yang telah dilakukan. Melalui tahapan tersebut diperoleh urutan nukleotida sampel yang positif diabetes dan sampel
FAKULTAS BIOLOGI LABORATORIUM GENETIKA & PEMULIAAN INSTRUKSI KERJA UJI
 Halaman : 1 dari 5 ISOLASI TOTAL DNA HEWAN DENGAN KIT EKSTRAKSI DNA 1. RUANG LINGKUP Metode ini digunakan untuk mengisolasi DNA dari sampel jaringan hewan, dapat dari insang, otot, darah atau jaringan
Halaman : 1 dari 5 ISOLASI TOTAL DNA HEWAN DENGAN KIT EKSTRAKSI DNA 1. RUANG LINGKUP Metode ini digunakan untuk mengisolasi DNA dari sampel jaringan hewan, dapat dari insang, otot, darah atau jaringan
HASIL DAN PEMBAHASAN. Uji Kualitatif dan Kuantitatif Hasil Isolasi RNA
 6 konsentrasinya. Untuk isolasi kulit buah kakao (outer pod wall dan inner pod wall) metode sama seperti isolasi RNA dari biji kakao. Uji Kualitatif dan Kuantitatif Hasil Isolasi RNA Larutan RNA hasil
6 konsentrasinya. Untuk isolasi kulit buah kakao (outer pod wall dan inner pod wall) metode sama seperti isolasi RNA dari biji kakao. Uji Kualitatif dan Kuantitatif Hasil Isolasi RNA Larutan RNA hasil
3 Metodologi Penelitian
 3 Metodologi Penelitian 3.1 Alat Penelitian ini dilakukan di Laboratorium Penelitian Biokimia, Program Studi Kimia, Institut Teknologi Bandung. Peralatan yang digunakan pada penelitian ini diantaranya
3 Metodologi Penelitian 3.1 Alat Penelitian ini dilakukan di Laboratorium Penelitian Biokimia, Program Studi Kimia, Institut Teknologi Bandung. Peralatan yang digunakan pada penelitian ini diantaranya
BAB III METODE PENELITIAN. Penelitian ini adalah penelitian deskriptif kualitatif untuk mengetahui
 BAB III METODE PENELITIAN 3.1 Rancangan Penelitian Penelitian ini adalah penelitian deskriptif kualitatif untuk mengetahui variasi genetik beberapa varietas mangga berdasarkan RAPD (Random Amplified Polymorphic
BAB III METODE PENELITIAN 3.1 Rancangan Penelitian Penelitian ini adalah penelitian deskriptif kualitatif untuk mengetahui variasi genetik beberapa varietas mangga berdasarkan RAPD (Random Amplified Polymorphic
BAB III METODE PENELITIAN. Penelitian ini merupakan penelitian yang dilakukan dengan metode
 16 BAB III METODE PENELITIAN A. Jenis Penelitian Penelitian ini merupakan penelitian yang dilakukan dengan metode deskriptif. Penelitian deskriptif adalah suatu metode penelitian untuk membuat deskripsi,
16 BAB III METODE PENELITIAN A. Jenis Penelitian Penelitian ini merupakan penelitian yang dilakukan dengan metode deskriptif. Penelitian deskriptif adalah suatu metode penelitian untuk membuat deskripsi,
Pengujian DNA, Prinsip Umum
 Pengujian DNA, Prinsip Umum Pengujian berbasis DNA dalam pengujian mutu benih memang saat ini belum diregulasikan sebagai salah satu standar kelulusan benih dalam proses sertifikasi. Dalam ISTA Rules,
Pengujian DNA, Prinsip Umum Pengujian berbasis DNA dalam pengujian mutu benih memang saat ini belum diregulasikan sebagai salah satu standar kelulusan benih dalam proses sertifikasi. Dalam ISTA Rules,
IDENTIFIKASI SPESIES POTYVIRUS
 TESIS IDENTIFIKASI SPESIES POTYVIRUS YANG BERASOSIASI DENGAN PENYAKIT MOSAIK PADA TANAMAN KACANG PANJANG (Vignasinensis L.) BERDASARKAN SEKUEN NUKLEOTIDA I WAYAN SUKADA PROGRAM PASCASARJANA UNIVERSITAS
TESIS IDENTIFIKASI SPESIES POTYVIRUS YANG BERASOSIASI DENGAN PENYAKIT MOSAIK PADA TANAMAN KACANG PANJANG (Vignasinensis L.) BERDASARKAN SEKUEN NUKLEOTIDA I WAYAN SUKADA PROGRAM PASCASARJANA UNIVERSITAS
Metodologi Penelitian. Metode, bahan dan alat yang digunakan dalam penelitian ini akan dipaparkan pada bab ini.
 Bab III Metodologi Penelitian Metode, bahan dan alat yang digunakan dalam penelitian ini akan dipaparkan pada bab ini. III.1 Rancangan Penelitian Secara garis besar tahapan penelitian dijelaskan pada diagram
Bab III Metodologi Penelitian Metode, bahan dan alat yang digunakan dalam penelitian ini akan dipaparkan pada bab ini. III.1 Rancangan Penelitian Secara garis besar tahapan penelitian dijelaskan pada diagram
3 METODOLOGI 3.1 Waktu dan Tempat 3.2 Bahan dan Alat
 3 METODOLOGI 3.1 Waktu dan Tempat Penelitian dilakukan selama bulan Januari hingga April 2010 bertempat di Laboratorium Karakteristik Bahan Baku, Departemen Teknologi Hasil Perairan, Fakultas Perikanan
3 METODOLOGI 3.1 Waktu dan Tempat Penelitian dilakukan selama bulan Januari hingga April 2010 bertempat di Laboratorium Karakteristik Bahan Baku, Departemen Teknologi Hasil Perairan, Fakultas Perikanan
BAB IV METODE PENELITIAN. Ruang lingkup dari penelitian ini meliputi bidang ilmu sitogenetika.
 BAB IV METODE PENELITIAN 4.1 Ruang lingkup penelitian Ruang lingkup dari penelitian ini meliputi bidang ilmu sitogenetika. 4.2 Tempat dan waktu penelitian Penelitian ini dilaksanakan di Pusat Riset Biomedik
BAB IV METODE PENELITIAN 4.1 Ruang lingkup penelitian Ruang lingkup dari penelitian ini meliputi bidang ilmu sitogenetika. 4.2 Tempat dan waktu penelitian Penelitian ini dilaksanakan di Pusat Riset Biomedik
Penelitian akan dilaksanakan pada bulan Februari-Agustus 2010 di Laboratorium Zoologi Departemen Biologi, FMIPA, IPB.
 Kolokium Ajeng Ajeng Siti Fatimah, Achmad Farajallah dan Arif Wibowo. 2009. Karakterisasi Genom Mitokondria Gen 12SrRNA - COIII pada Ikan Belida Batik Anggota Famili Notopteridae. Kolokium disampaikan
Kolokium Ajeng Ajeng Siti Fatimah, Achmad Farajallah dan Arif Wibowo. 2009. Karakterisasi Genom Mitokondria Gen 12SrRNA - COIII pada Ikan Belida Batik Anggota Famili Notopteridae. Kolokium disampaikan
II. BAHAN DAN METODE
 II. BAHAN DAN METODE 2.1. Waktu dan Lokasi Penelitian Penelitian ini dilaksanakan dari bulan Agustus sampai September tahun 2011. Sampel ikan berasal dari 3 lokasi yaitu Jawa (Jawa Barat), Sumatera (Jambi),
II. BAHAN DAN METODE 2.1. Waktu dan Lokasi Penelitian Penelitian ini dilaksanakan dari bulan Agustus sampai September tahun 2011. Sampel ikan berasal dari 3 lokasi yaitu Jawa (Jawa Barat), Sumatera (Jambi),
BAHAN DAN METODE. Analisis Kekerabatan Rayap Tanah M. gilvus dengan Pendekatan Perilaku
 BAHAN DAN METODE Tempat dan Waktu Sampel rayap diambil dari Cagar Alam Yanlappa-Jasinga dan Kampus IPB- Dramaga, Bogor. Rayap diidentifikasi dan diuji perilaku agonistiknya di Laboratorium Biosistematika
BAHAN DAN METODE Tempat dan Waktu Sampel rayap diambil dari Cagar Alam Yanlappa-Jasinga dan Kampus IPB- Dramaga, Bogor. Rayap diidentifikasi dan diuji perilaku agonistiknya di Laboratorium Biosistematika
LAPORAN PRAKTIKUM REKAYASA GENETIKA
 LAPORAN PRAKTIKUM REKAYASA GENETIKA LAPORAN IV (ISOLASI RNA DARI TANAMAN) KHAIRUL ANAM P051090031/BTK BIOTEKNOLOGI SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR 2010 0 ISOLASI RNA DARI TANAMAN TUJUAN Tujuan
LAPORAN PRAKTIKUM REKAYASA GENETIKA LAPORAN IV (ISOLASI RNA DARI TANAMAN) KHAIRUL ANAM P051090031/BTK BIOTEKNOLOGI SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR 2010 0 ISOLASI RNA DARI TANAMAN TUJUAN Tujuan
Lampiran 1 Ekstraksi dan isolasi DNA dengan metode GeneAid
 LAMPIRAN 9 Lampiran 1 Ekstraksi dan isolasi DNA dengan metode GeneAid Satu ruas tungkai udang mantis dalam etanol dipotong dan dimasukkan ke dalam tube 1,5 ml. Ruas tungkai yang telah dipotong (otot tungkai)
LAMPIRAN 9 Lampiran 1 Ekstraksi dan isolasi DNA dengan metode GeneAid Satu ruas tungkai udang mantis dalam etanol dipotong dan dimasukkan ke dalam tube 1,5 ml. Ruas tungkai yang telah dipotong (otot tungkai)
4 HASIL DAN PEMBAHASAN
 4 HASIL DAN PEMBAHASAN 4.1 Karakteristik Morfologi Pada penelitian ini digunakan lima sampel koloni karang yang diambil dari tiga lokasi berbeda di sekitar perairan Kepulauan Seribu yaitu di P. Pramuka
4 HASIL DAN PEMBAHASAN 4.1 Karakteristik Morfologi Pada penelitian ini digunakan lima sampel koloni karang yang diambil dari tiga lokasi berbeda di sekitar perairan Kepulauan Seribu yaitu di P. Pramuka
4 KISARAN INANG Chili veinal mottle virus ISOLAT LEMAH (Host Range Study of Weak Isolates of Chili veinal mottle virus)
 22 4 KISARAN INANG Chili veinal mottle virus ISOLAT LEMAH (Host Range Study of Weak Isolates of Chili veinal mottle virus) Abstrak Chili veinal mottle virus (ChiVMV) merupakan salah satu penyakit penting
22 4 KISARAN INANG Chili veinal mottle virus ISOLAT LEMAH (Host Range Study of Weak Isolates of Chili veinal mottle virus) Abstrak Chili veinal mottle virus (ChiVMV) merupakan salah satu penyakit penting
III. MATERI DAN METODE. Fakultas Pertanian dan Peternakan Universitas Islam Negeri Sultan Syarif Kasim
 III. MATERI DAN METODE 3.1 Waktu dan Tempat Penelitian ini akan dilaksanakan di Laboratorium Genetika dan Pemuliaan Fakultas Pertanian dan Peternakan Universitas Islam Negeri Sultan Syarif Kasim Riau Pekanbaru
III. MATERI DAN METODE 3.1 Waktu dan Tempat Penelitian ini akan dilaksanakan di Laboratorium Genetika dan Pemuliaan Fakultas Pertanian dan Peternakan Universitas Islam Negeri Sultan Syarif Kasim Riau Pekanbaru
BAHAN DAN METODE. Tahapan Analisis DNA S. incertulas
 11 BAHAN DAN METODE Waktu dan Tempat Penelitian Penelitian ini dilaksanakan pada bulan Juli 2010 sampai dengan Mei 2011. Koleksi sampel dilakukan pada beberapa lokasi di Jawa Tengah, Daerah Istimewa Yogyakarta
11 BAHAN DAN METODE Waktu dan Tempat Penelitian Penelitian ini dilaksanakan pada bulan Juli 2010 sampai dengan Mei 2011. Koleksi sampel dilakukan pada beberapa lokasi di Jawa Tengah, Daerah Istimewa Yogyakarta
IDENTIFIKASI SPESIES POLEROVIRUS PENYEBAB PENYAKIT KLOROSIS PADA TANAMAN CABAI MELALUI SEKUEN NUKLEOTIDA TITIN RAHAYUNINGSIH
 1 IDENTIFIKASI SPESIES POLEROVIRUS PENYEBAB PENYAKIT KLOROSIS PADA TANAMAN CABAI MELALUI SEKUEN NUKLEOTIDA TITIN RAHAYUNINGSIH DEPARTEMEN PROTEKSI TANAMAN FAKULTAS PERTANIAN INSTITUT PERTANIAN BOGOR BOGOR
1 IDENTIFIKASI SPESIES POLEROVIRUS PENYEBAB PENYAKIT KLOROSIS PADA TANAMAN CABAI MELALUI SEKUEN NUKLEOTIDA TITIN RAHAYUNINGSIH DEPARTEMEN PROTEKSI TANAMAN FAKULTAS PERTANIAN INSTITUT PERTANIAN BOGOR BOGOR
4 HASIL DAN PEMBAHASAN
 4 HASIL DAN PEMBAHASAN 4.1 Preparasi dan Karakteristik Bahan Baku Produk tuna steak dikemas dengan plastik dalam keadaan vakum. Pengemasan dengan bahan pengemas yang cocok sangat bermanfaat untuk mencegah
4 HASIL DAN PEMBAHASAN 4.1 Preparasi dan Karakteristik Bahan Baku Produk tuna steak dikemas dengan plastik dalam keadaan vakum. Pengemasan dengan bahan pengemas yang cocok sangat bermanfaat untuk mencegah
BAB III METODE PENELITIAN
 BAB III METODE PENELITIAN A. Jenis Penelitian Jenis penelitian yang digunakan adalah penelitian dasar dengan metode deskriptif (Nazir, 1983). B. Populasi dan Sampel Populasi dalam penelitian ini adalah
BAB III METODE PENELITIAN A. Jenis Penelitian Jenis penelitian yang digunakan adalah penelitian dasar dengan metode deskriptif (Nazir, 1983). B. Populasi dan Sampel Populasi dalam penelitian ini adalah
VISUALISASI HASIL PCR DENGAN METODE PCR LANGSUNG DAN TIDAK LANGSUNG PADA SAMPEL BAKTERI Pseudomonas fluorescens dan Ralstonia solanacearum
 VISUALISASI HASIL PCR DENGAN METODE PCR LANGSUNG DAN TIDAK LANGSUNG PADA SAMPEL BAKTERI Pseudomonas fluorescens dan Ralstonia solanacearum Pendahuluan Polymerase Chain Reaction (PCR) adalah suatu teknik
VISUALISASI HASIL PCR DENGAN METODE PCR LANGSUNG DAN TIDAK LANGSUNG PADA SAMPEL BAKTERI Pseudomonas fluorescens dan Ralstonia solanacearum Pendahuluan Polymerase Chain Reaction (PCR) adalah suatu teknik
MATERI DAN METODE. Lokasi dan Waktu. Materi
 MATERI DAN METODE Lokasi dan Waktu Penelitian ini dilaksanakan di Laboratorium Genetika Molekuler, Bagian Pemuliaan dan Genetika Ternak, Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan,
MATERI DAN METODE Lokasi dan Waktu Penelitian ini dilaksanakan di Laboratorium Genetika Molekuler, Bagian Pemuliaan dan Genetika Ternak, Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan,
Pengaruh Infeksi Beberapa Jenis Virus terhadap Penurunan Hasil pada Tanaman Cabai Rawit (Capsicum frutescens L.)
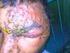 Pengaruh Infeksi Beberapa Jenis Virus terhadap Penurunan Hasil pada Tanaman Cabai Rawit (Capsicum frutescens L.) I WAYAN SUKADA I MADE SUDANA I DEWA NYOMAN NYANA *) GEDE SUASTIKA KETUT SIADI 1 1 Program
Pengaruh Infeksi Beberapa Jenis Virus terhadap Penurunan Hasil pada Tanaman Cabai Rawit (Capsicum frutescens L.) I WAYAN SUKADA I MADE SUDANA I DEWA NYOMAN NYANA *) GEDE SUASTIKA KETUT SIADI 1 1 Program
VII. PEMBAHASAN UMUM
 VII. PEMBAHASAN UMUM Tanaman cabai (Capsicum annuum) merupakan salah satu komoditas andalan hortikultura di Indonesia. Tanaman tersebut ditanam di seluruh provinsi di Indonesia dan memiliki nilai ekonomis
VII. PEMBAHASAN UMUM Tanaman cabai (Capsicum annuum) merupakan salah satu komoditas andalan hortikultura di Indonesia. Tanaman tersebut ditanam di seluruh provinsi di Indonesia dan memiliki nilai ekonomis
BAB III BAHAN DAN METODE
 BAB III BAHAN DAN METODE 3.1. Tempat dan Waktu Penelitian 3.1.1. Tempat Penelitian Penelitian dilaksanakan di Laboratorium Bioteknologi Perikanan dan Kelautan Fakultas Perikanan dan Ilmu Kelautan Universitas
BAB III BAHAN DAN METODE 3.1. Tempat dan Waktu Penelitian 3.1.1. Tempat Penelitian Penelitian dilaksanakan di Laboratorium Bioteknologi Perikanan dan Kelautan Fakultas Perikanan dan Ilmu Kelautan Universitas
III. METODE PENELITIAN. Wajwalku Wildlife Laboratory, Fakultas Kedokteran Hewan, Universitas Kasetsart
 III. METODE PENELITIAN A. Waktu dan Lokasi Penelitian Penelitian dilakukan pada bulan Februari hingga Maret 2016. Preparasi penelitian ini dilakukan di Laboratorium Biologi Molekuler, Fakultas Teknobiologi
III. METODE PENELITIAN A. Waktu dan Lokasi Penelitian Penelitian dilakukan pada bulan Februari hingga Maret 2016. Preparasi penelitian ini dilakukan di Laboratorium Biologi Molekuler, Fakultas Teknobiologi
BAB III METODOLOGI PENELITIAN. Jenis penelitian yang digunakan adalah penelitian dasar dengan metode
 24 BAB III METODOLOGI PENELITIAN A. Jenis Penelitian Jenis penelitian yang digunakan adalah penelitian dasar dengan metode penelitian deskriptif. B. Objek Penelitian Empat spesies burung anggota Famili
24 BAB III METODOLOGI PENELITIAN A. Jenis Penelitian Jenis penelitian yang digunakan adalah penelitian dasar dengan metode penelitian deskriptif. B. Objek Penelitian Empat spesies burung anggota Famili
DETEKSI SIMULTAN CMV DAN CHIVMV PENYEBAB PENYAKIT MOSAIK PADA TANAMAN CABAI DENGAN DUPLEX RT-PCR. Udayana. Jl. PB. Sudirman Denpasar Bali Indonesia
 DETEKSI SIMULTAN CMV DAN CHIVMV PENYEBAB PENYAKIT MOSAIK PADA TANAMAN CABAI DENGAN DUPLEX RT-PCR I Gede Agus Adi Chandra 1, I Dewa Nyoman Nyana 2*, I G N Alit Susanta Wirya 2, Gede Suastika 3 1 Program
DETEKSI SIMULTAN CMV DAN CHIVMV PENYEBAB PENYAKIT MOSAIK PADA TANAMAN CABAI DENGAN DUPLEX RT-PCR I Gede Agus Adi Chandra 1, I Dewa Nyoman Nyana 2*, I G N Alit Susanta Wirya 2, Gede Suastika 3 1 Program
BAB IV HASIL DAN PEMBAHASAN. telah banyak dilakukan sebelumnya menunjukkan bahwa fenomena munculnya
 BAB IV HASIL DAN PEMBAHASAN Penelitian terhadap urutan nukleotida daerah HVI mtdna manusia yang telah banyak dilakukan sebelumnya menunjukkan bahwa fenomena munculnya rangkaian poli-c merupakan fenomena
BAB IV HASIL DAN PEMBAHASAN Penelitian terhadap urutan nukleotida daerah HVI mtdna manusia yang telah banyak dilakukan sebelumnya menunjukkan bahwa fenomena munculnya rangkaian poli-c merupakan fenomena
Abstrak. Kata kunci : Chili veinal mottle virus, isolat lemah, tanaman cabai. Abstract
 3 3 EKSPLORASI Chili veinal mottle virus ISOLAT LEMAH DARI PERTAMAN CABAI (Exploration of Weak Isolates of Chili veinal mottle virus from Chili Pepper) Abstrak Salah satu virus utama yang menginfeksi tanaman
3 3 EKSPLORASI Chili veinal mottle virus ISOLAT LEMAH DARI PERTAMAN CABAI (Exploration of Weak Isolates of Chili veinal mottle virus from Chili Pepper) Abstrak Salah satu virus utama yang menginfeksi tanaman
E-Jurnal Agroekoteknologi Tropika ISSN: Vol. 4, No. 1, Januari 2015
 Deteksi Cucumber Mosaic Virus (CMV) dan Chili Veinal Mottle Virus (ChiVMV) pada Gulma Commelina spp. di Pertanaman Cabai (Capsicum spp.) Melalui Teknik Uji Serologi dan Molekuler NI KADEK VENIARI 1 KETUT
Deteksi Cucumber Mosaic Virus (CMV) dan Chili Veinal Mottle Virus (ChiVMV) pada Gulma Commelina spp. di Pertanaman Cabai (Capsicum spp.) Melalui Teknik Uji Serologi dan Molekuler NI KADEK VENIARI 1 KETUT
BIOMA, Juni 2014 ISSN: Vol. 16, No. 1, Hal
 BIOMA, Juni 2014 ISSN: 1410-8801 Vol. 16, No. 1, Hal. 18-25 Karakterisasi Genetik Fragmen Gen Penyandi RNA Polimerase Infectious Myonecrosis Virus (IMNV) yang Menginfeksi Udang Vannamei (Litopenaeus vannamei
BIOMA, Juni 2014 ISSN: 1410-8801 Vol. 16, No. 1, Hal. 18-25 Karakterisasi Genetik Fragmen Gen Penyandi RNA Polimerase Infectious Myonecrosis Virus (IMNV) yang Menginfeksi Udang Vannamei (Litopenaeus vannamei
DAFTAR ISI Halaman HALAMAN JUDUL... i HALAMAN PENGESAHAN... KATA PENGANTAR... DAFTAR ISI... DAFTAR TABEL... DAFTAR GAMBAR... DAFTAR LAMPIRAN...
 DAFTAR ISI HALAMAN JUDUL... i HALAMAN PENGESAHAN... KATA PENGANTAR...... DAFTAR ISI... DAFTAR TABEL... DAFTAR GAMBAR... DAFTAR LAMPIRAN... INTISARI... ABSTRACT... PENDAHULUAN... 1 A. Latar Belakang...
DAFTAR ISI HALAMAN JUDUL... i HALAMAN PENGESAHAN... KATA PENGANTAR...... DAFTAR ISI... DAFTAR TABEL... DAFTAR GAMBAR... DAFTAR LAMPIRAN... INTISARI... ABSTRACT... PENDAHULUAN... 1 A. Latar Belakang...
4. HASIL DAN PEMBAHASAN
 29 4. HASIL DAN PEMBAHASAN 4.1 Karakteristik isolat bakteri dari ikan tuna dan cakalang 4.1.1 Morfologi isolat bakteri Secara alamiah, mikroba terdapat dalam bentuk campuran dari berbagai jenis. Untuk
29 4. HASIL DAN PEMBAHASAN 4.1 Karakteristik isolat bakteri dari ikan tuna dan cakalang 4.1.1 Morfologi isolat bakteri Secara alamiah, mikroba terdapat dalam bentuk campuran dari berbagai jenis. Untuk
METODOLOGI. Gambar 1 Bahan tanaman : (a) Tetua IR64; (b) tetua Hawarabunar, dan (c) F 1 (IRxHawarabunar) c a b
 METODOLOGI Waktu dan Tempat Penelitian ini dilakukan dua tahap yaitu penanaman padi dan analisis fisiologi dan marka molekuler. Penanaman padi secara gogo pada tanah masam dilakukan di rumah kaca Cikabayan
METODOLOGI Waktu dan Tempat Penelitian ini dilakukan dua tahap yaitu penanaman padi dan analisis fisiologi dan marka molekuler. Penanaman padi secara gogo pada tanah masam dilakukan di rumah kaca Cikabayan
ANALISIS KERAGAMAN GENETIK MUTAN JARAK PAGAR (Jatropha curcas L.) HASIL PERLAKUAN MUTAGEN KOLKISIN BERDASARKAN PENANDA MOLEKULER RAPD
 ANALISIS KERAGAMAN GENETIK MUTAN JARAK PAGAR (Jatropha curcas L.) HASIL PERLAKUAN MUTAGEN KOLKISIN BERDASARKAN PENANDA MOLEKULER RAPD Herdiyana Fitriani Dosen Program Studi Pendidikan Biologi FPMIPA IKIP
ANALISIS KERAGAMAN GENETIK MUTAN JARAK PAGAR (Jatropha curcas L.) HASIL PERLAKUAN MUTAGEN KOLKISIN BERDASARKAN PENANDA MOLEKULER RAPD Herdiyana Fitriani Dosen Program Studi Pendidikan Biologi FPMIPA IKIP
BAHAN DAN METODE Tempat dan Waktu Bahan dan Alat Metode Penelitian Isolasi Aktinomiset
 BAHAN DAN METODE Tempat dan Waktu Penelitian dilaksanakan di Laboratorium Bakteriologi Tumbuhan, Departemen Proteksi Tanaman, Fakultas Pertanian, Institut Pertanian Bogor, dari bulan Februari sampai dengan
BAHAN DAN METODE Tempat dan Waktu Penelitian dilaksanakan di Laboratorium Bakteriologi Tumbuhan, Departemen Proteksi Tanaman, Fakultas Pertanian, Institut Pertanian Bogor, dari bulan Februari sampai dengan
HASIL DAN PEMBAHASAN. DNA Genom
 IV. HASIL DAN PEMBAHASAN A. Isolasi DNA Metode isolasi dilakukan untuk memisahkan DNA dari komponen sel yang lain (Ilhak dan Arslan, 2007). Metode isolasi ini sesuai dengan protokol yang diberikan oleh
IV. HASIL DAN PEMBAHASAN A. Isolasi DNA Metode isolasi dilakukan untuk memisahkan DNA dari komponen sel yang lain (Ilhak dan Arslan, 2007). Metode isolasi ini sesuai dengan protokol yang diberikan oleh
BAB III BAHAN DAN METODE PENELITIAN
 BAB III BAHAN DAN METODE PENELITIAN 3.1 Waktu dan Lokasi Penelitian 3.1.1. Lokasi Penelitian Penelitian ini dilakukan di Laboratorium Bioteknologi Perikanan dan Kelautan Fakultas Perikanan dan Ilmu Kelautan
BAB III BAHAN DAN METODE PENELITIAN 3.1 Waktu dan Lokasi Penelitian 3.1.1. Lokasi Penelitian Penelitian ini dilakukan di Laboratorium Bioteknologi Perikanan dan Kelautan Fakultas Perikanan dan Ilmu Kelautan
BAB III METODE PENELITIAN
 BAB III METODE PENELITIAN 3.1 Rancangan Penelitian Penelitian tentang Karakterisasi genetik Udang Jari (Metapenaeus elegans De Man, 1907) hasil tangkapan dari Laguna Segara Anakan berdasarkan haplotipe
BAB III METODE PENELITIAN 3.1 Rancangan Penelitian Penelitian tentang Karakterisasi genetik Udang Jari (Metapenaeus elegans De Man, 1907) hasil tangkapan dari Laguna Segara Anakan berdasarkan haplotipe
HASIL DAN PEMBAHASAN
 HASIL DAN PEMBAHASAN Identitas Kutukebul Pengkoloni Pertanaman Tomat Kutukebul yang dikumpulkan dari pertanaman tomat di daerah Cisarua, Bogor diperbanyak di tanaman tomat dalam kurungan kedap serangga
HASIL DAN PEMBAHASAN Identitas Kutukebul Pengkoloni Pertanaman Tomat Kutukebul yang dikumpulkan dari pertanaman tomat di daerah Cisarua, Bogor diperbanyak di tanaman tomat dalam kurungan kedap serangga
BAB 3 METODOLOGI PENELITIAN
 20 BAB 3 METODOLOGI PENELITIAN 3.1. Desain Penelitian Penelitian ini bersifat deskriptif cross sectional molekuler. Data yang diperoleh berasal dari pemeriksaan langsung yang dilakukan peneliti sebanyak
20 BAB 3 METODOLOGI PENELITIAN 3.1. Desain Penelitian Penelitian ini bersifat deskriptif cross sectional molekuler. Data yang diperoleh berasal dari pemeriksaan langsung yang dilakukan peneliti sebanyak
II. BAHAN DAN METODE. Betina BEST BB NB RB. Nirwana BN NN RN. Red NIFI BR NR RR
 II. BAHAN DAN METODE Ikan Uji Ikan uji yang digunakan adalah ikan nila hibrida hasil persilangan resiprok 3 strain BEST, Nirwana dan Red NIFI koleksi Balai Riset Perikanan Budidaya Air Tawar Sempur, Bogor.
II. BAHAN DAN METODE Ikan Uji Ikan uji yang digunakan adalah ikan nila hibrida hasil persilangan resiprok 3 strain BEST, Nirwana dan Red NIFI koleksi Balai Riset Perikanan Budidaya Air Tawar Sempur, Bogor.
bio.unsoed.ac.id METODE PENELITIAN A. Materi, Lokasi, dan Waktu Penelitian 1. Materi Penelitian
 III. METODE PENELITIAN A. Materi, Lokasi, dan Waktu Penelitian 1. Materi Penelitian 1.1. Peralatan Penelitian Alat yang digunakan dalam penelitian ini adalah botol sampel, beaker glass, cool box, labu
III. METODE PENELITIAN A. Materi, Lokasi, dan Waktu Penelitian 1. Materi Penelitian 1.1. Peralatan Penelitian Alat yang digunakan dalam penelitian ini adalah botol sampel, beaker glass, cool box, labu
TEMUAN PENYAKIT BARU. Tomato yellow leaf curl Kanchanaburi virus Penyebab Penyakit Mosaik Kuning pada Tanaman Terung di Jawa
 ISSN: 2339-2479 Volume 9, Nomor 4, Agustus 2013 Halaman 127-131 DOI: 10.14692/jfi.9.4.127 TEMUAN PENYAKIT BARU Tomato yellow leaf curl Kanchanaburi virus Penyebab Penyakit Mosaik Kuning pada Tanaman Terung
ISSN: 2339-2479 Volume 9, Nomor 4, Agustus 2013 Halaman 127-131 DOI: 10.14692/jfi.9.4.127 TEMUAN PENYAKIT BARU Tomato yellow leaf curl Kanchanaburi virus Penyebab Penyakit Mosaik Kuning pada Tanaman Terung
HASIL DAN PEMBAHASAN Pengujian Kuantitas dan Kualitas DNA
 HASIL DAN PEMBAHASAN Gen sitokrom b digunakan sebagai pembawa kode genetik seperti halnya gen yang terdapat dalam nukleus. Primer tikus yang dikembangkan dari gen sitokrom b, terbukti dapat mengamplifikasi
HASIL DAN PEMBAHASAN Gen sitokrom b digunakan sebagai pembawa kode genetik seperti halnya gen yang terdapat dalam nukleus. Primer tikus yang dikembangkan dari gen sitokrom b, terbukti dapat mengamplifikasi
2015 ISOLASI DNA PARSIAL GEN
 1 BAB I PENDAHULUAN A. Latar Belakang Penelitian Ikan gurame (Osphronemus merupakan salah satu ikan air tawar yang termasuk ke dalam infraclass Teleostei (Integrated Taxonomic Information System, 2012).
1 BAB I PENDAHULUAN A. Latar Belakang Penelitian Ikan gurame (Osphronemus merupakan salah satu ikan air tawar yang termasuk ke dalam infraclass Teleostei (Integrated Taxonomic Information System, 2012).
MATERI DAN METODE. Materi. Tabel 1. Sampel Darah Sapi Perah dan Sapi Pedaging yang Digunakan No. Bangsa Sapi Jenis Kelamin
 MATERI DAN METODE Lokasi dan Waktu Penelitian ini dilaksanakan di Laboratorium Genetika Molekuler Ternak, Bagian Pemuliaan dan Genetika, Fakultas Peternakan, Institut Pertanian Bogor. Penelitian ini berlangsung
MATERI DAN METODE Lokasi dan Waktu Penelitian ini dilaksanakan di Laboratorium Genetika Molekuler Ternak, Bagian Pemuliaan dan Genetika, Fakultas Peternakan, Institut Pertanian Bogor. Penelitian ini berlangsung
Deteksi Molekuler Cucumber Mosaic Virus (Cmv) pada Tanaman Gamal (Gliricidia Sepium) Sebagai Barier pada Pertanaman Cabai
 Deteksi Molekuler Cucumber Mosaic Virus (Cmv) pada Tanaman Gamal (Gliricidia Sepium) Sebagai Barier pada Pertanaman Cabai IDA BAGUS GDE PRANATAYANA I GEDE RAI MAYA TEMAJA*) KETUT AYU YULIADHI 1 I DEWA
Deteksi Molekuler Cucumber Mosaic Virus (Cmv) pada Tanaman Gamal (Gliricidia Sepium) Sebagai Barier pada Pertanaman Cabai IDA BAGUS GDE PRANATAYANA I GEDE RAI MAYA TEMAJA*) KETUT AYU YULIADHI 1 I DEWA
IDENTIFIKASI MOLEKULER BEAN COMMON MOSAIC VIRUS YANG BERASOSIASI DENGAN PENYAKIT MOSAIK KUNING KACANG PANJANG
 J. HPT Tropika. ISSN 1411-7525 132 J. HPT Tropika Vol. 15 No. 2, 2015: 132-140 Vol. 15, No. 2: 132 140, September 2015 IDENTIFIKASI MOLEKULER BEAN COMMON MOSAIC VIRUS YANG BERASOSIASI DENGAN PENYAKIT MOSAIK
J. HPT Tropika. ISSN 1411-7525 132 J. HPT Tropika Vol. 15 No. 2, 2015: 132-140 Vol. 15, No. 2: 132 140, September 2015 IDENTIFIKASI MOLEKULER BEAN COMMON MOSAIC VIRUS YANG BERASOSIASI DENGAN PENYAKIT MOSAIK
OPTIMASI PCR (Polymerase Chain Reaction) FRAGMEN 724 pb GEN katg MULTI DRUG RESISTANCE TUBERCULOSIS UNTUK MENINGKATKAN PRODUK AMPLIFIKASI
 OPTIMASI PCR (Polymerase Chain Reaction) FRAGMEN 724 pb GEN katg MULTI DRUG RESISTANCE TUBERCULOSIS UNTUK MENINGKATKAN PRODUK AMPLIFIKASI Deniariasih, N.W. 1, Ratnayani, K. 2, Yowani, S.C. 1 1 Jurusan
OPTIMASI PCR (Polymerase Chain Reaction) FRAGMEN 724 pb GEN katg MULTI DRUG RESISTANCE TUBERCULOSIS UNTUK MENINGKATKAN PRODUK AMPLIFIKASI Deniariasih, N.W. 1, Ratnayani, K. 2, Yowani, S.C. 1 1 Jurusan
